Sturktur der Nukleinsäuren
Die Denaturierung der DNA kann man durch eine Veränderung der
Brechungseigenschaften für UV-Licht darstellen. Bei der Denaturierung
kommt es zu einer Hyperchromatie, einem Anstieg der optischen
Dichte. Der Punkt, an dem sich die Stränge trennen wird Tm
genannt. Der Schmelzpunkt wird durch einen hohen GC-Gehalt (drei
Wasserstoffbrücken) nach oben hin verschoben.
Nachdem die DNA denaturiert wurde, kann sie unter geeigneten
Bedinungen auch wieder renaturieren.
Wenn man unterschiedliche DNA-Einzelstränge dazu bringt, sich zu
reassoziieren, spricht man von einer Hybridisierung. Diese ist ein
exaktes Mass für die Komplementarität der Einzelstränge.
Bei einer Hybridisierung in Lösung verfolgt man den Verlauf entweder
an Hand der optischen Dichte oder an Hand des Einbaus eines
radioaktiven Markers in die DNA. Da diese Methode allerdings relativ
ungenau ist, verwendet man in der Praxis zumeist die
Filterhybridisierung, bei der einer der Stränge an eine
cellulsemembran gebunden ist man dann die Bindung der anderen DNA an
die fixierte misst.
Die Energie, die benütigt wird (oder frei wird), damit sich zwei
DNA-Regionen paaren, lässt sich berechenen, indem man die 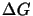 -Werte die zur Bildung oder Aufrechterhaltung der Paarung nötig
sind, aufaddiert. Dabei ist - auf Grund von hydrophoben
Wechselwirkungen - auch die Abfolge der Basenpaarduos entscheidend. -Werte die zur Bildung oder Aufrechterhaltung der Paarung nötig
sind, aufaddiert. Dabei ist - auf Grund von hydrophoben
Wechselwirkungen - auch die Abfolge der Basenpaarduos entscheidend.
In vitro findet man neben der helicalen Struktur auch die
Haarnadelstruktur, die sich bei innerhalb eines Strangs Sequenzen
zusammenlagern, Blasen, die entstehen, wenn eine Sequen keinen
Gegenpart auf dem anderen Strang hat und Innere Schleifen, die dann
entstehen, wenn die beiden Stänge keine Basenpaarung miteinandern
eingehen.
Auch in vivo findet man bei der DNA unterschiedliche
Doppelhelixstrukturen. Die übliche Form der DNA ist die
B-Konformation, die durch eine grosse und eine kleine Furche. Die
Anzahl der Basen pro Windung liegt bei 10,4 und entsprechend liegt der
Winkel zwischen zwei Basen bei 34,6. Innerhalb der Familie von
B-Strukturen kann es zu leichten Abweichungen von diesen Werten
kommen.
Die A-Konformation ist der doppelsträngigen Form der RNA sehr ähnlich,
da diese - mangels Hydroxygruppe - keine A-Konformation ausbilden
kann. Bei dieser Form findet man mehr Basenpaare pro Windung. Die
Struktur ist ,,dichter`` und die grosse Furche weniger gut
zugänglich.
Die Z-Konformation bildet eine linksgewundene Doppelhelix aus. Sie hat
die meisten Basenpaar pro Windung und ist die dünnste Struktur, bei
der das Rückgrat in einem zick-zack entlang der Helix verläuft. Eine
Bildung dieser Konformation in vivo ist unwahrscheinlich.
Bei den ringförmigne DNA-Molekülen findet man eine Form der
Überspiralisierung (supercoiling). So kann eine superhelicale Struktur
entstehen. Bei einer negaiven Überspiralisierung (unterdreht) kann es
dazu kommen, dass die Spannung abgebaut wird, indem in begrenztem
Umfang Basenpaare gelöst werden.
Man findet in vivo häufig eine negative Überspiralisierung, da diese
als eine Art Energiespeicher dienen kann, der bei der Trennung der DNA
als Energeiquelle dient.
|





