Unterabschnitte
Kontrolle der Genexpression
Alle Zellen haben die gleiche DNA und auch ein grosser Anteil an
Proteinen weicht zwischen den Zellen nur wenig ab. Dies sind z.B. die
Proteine der Proteinbiosynthese, des Cytoskeletts, des ER, des Golgi,
etc.
Die Verschiedenheit der Zellen kommt durch differentielle
Genexpression zu stande.
Die Genexpression kann auf unterschiedlichen Stufen kontrolliert
werden:
- Durch Transkriptionskontrolle wird kontrolliert wann und wie oft
ein Gen transkribiert wird.
- Durch RNA-Processing-Kontrolle wird kontrolliert, wie das
Splicen vor sich geht.
- Durch Kontrolle des RNA-Transportes wird kontolliert, welche RNA
den Zellkern verlässt.
- Durch Translationskontrolle wird die Translation der Proteine
gesteuert.
- Durch die Kontrolle des mRNA-Abbaus können bestimmte RNAs
selektiv destabilsiert werden.
- Durch Aktivierung oder Inaktivierung können die Proteine auch
nach ihrer Translation noch moduliert werden.
Regulatorproteine
Ein Promotor ist eine DNA-Sequenz, die die RNA-Polymerase dazu bringt,
an der geöffneten DNA an die Startsequenz zu binden.
Innerhalb der Promotorsequenz liegt der Operator, eine spezifische
Sequenz, an die z.B. das HTH-Motiv eines regulatorischen Proteins
binden kann. Wenn dieses Regulatorprotein - der Repressor - an den
Operator bindet, kann die RNA-Polymerase nicht binden und die Sequenz
nicht transkribiert werden. Der Repressor wird zumeist in einem
Feedback von dem Produkt der Transkription so verändert, dass er
an die DNA binden kann. Dieser Mechanismus wird als negative
Regulation bezeichnet.
Im Gegensatz zu einem Repressor aktiviert der Aktivator die
Transkription. Bei dieser positiven Regulation bindet ein - meist
ebenfalls Ligandenabhängiges - Protein an die DNA und aktiviert
dadurch die Transkription des Gens.
Es gibt einige Proteine, die je nach Lage ihrer Bindungsstelle
entweder als Aktivator oder als Repressor fungieren.
Bei komplexeren Schaltern, wir z.B. beim lac-Operon findet man
Kombinationen aus Repressor und Aktivator.
Die lac-Proteine transportieren Lactose in die Zelle und bauen sie
ab. Ist keine Lactose vorhanden, bindet ein lac-Repressor und
verhindert die Transkription des Gens. Nur wenn Lactose vorhanden ist
und der keine Glucose vorhanden ist, kann das Gen abgelesen werden, da
nur dann auch ein Aktivator bindet, der die Transkription ermöglicht.
Die Transkriptionskontrolle bei Eukaryonten ist wesentlich komplexer
als dieses einfache Beispiel aus E. coli.
Bei Eukaryonten unterscheidet sich der Aufbau vor allem dadurch, dass
zum einen Promotoren sich in grossem Abstand zur eigentlich
regulierten Sequenz befinden können und zum anderen eine Gruppe von
allgemeinen Transkriptionsfaktoren für den Beginn der Transkription
notwendig ist.
Die allgemeinen Transkriptinsfaktoren müssen sich - damit die
Transkription beginnen kann - in einer bestimmten Reihenfolge an die
DNA anlagern. Erst so wird die Bindung und Funktion der RNA-Polymerase
ermöglicht.
Zunächst bindet der sogenannte TATA-Faktor an die TATA-Box, eine kurze
doppel-hlicale Sequenz, die vor allem aus T und A besteht. Diese Box
liegt normalerweise ca. 25 Basen stromaufwärts vom Startpunkt der
Transkription. Dieser Faktor besteht aus mehreren Untereinheiten. Wenn
diese an die DNA gebunden haben, dann folgen die anderen Elemente und
die RNA-Polymerase rasch nach.
Dadurch, dass mehrere Schritte bis zum Beginn der Transkription
notwendig sind, ist dieser Prozess auf mehreren Ebenen und sehr genau
zu regulieren.
Eine andere Möglichkeit, die Transkription zu beeinflussen besteht in
einer Bindung von Regulatorproteinen an die sogenannten Enhancer. Das
besondere an der Wirkung ist, dass sich die Enhancer weit von dem
eigentlichen Gen entfernt befinden können.
Sie wirken auf die Transkription, indem sie an den den Enhancer binden
und die DNA dann eine Schleife bindet, durch die das am Enhancer
gebundene Protein mit den am Promotor gebundenen Proteinen oder der
Polymerase selbst wechselwirken kann. Durch die DNA wird die Anzahl
der Interaktionen zwischen Enhancer- und Promotorregion räumlich
gekoppelt und dadurch wahrscheinlicher.
Die Gen-Kontrollregion umfasst alle an der Transkriptionskontrolle
beteiligten Sequenzen. Es haben sich bei der Transkriptionskontrolle
der Eukaryonten viele verschiedene Mechanismen herausgebildet.
Die einfachen Aktivatorproteine bestehen zumeist aus zwei
Untereinheiten. Diese eine DNA-bindende Domäne bindet an einer
spezifischen Bindungsstelle, während die Aktivierungsdomäne
z.B. Kontakt mit der Polymerase aufnimmt und so die Transkription
beschleunigt. Diesen Mechanismus konnte man mit Fusionsproteinen aus
unterschiedlichen Aktivator- und DNA-bindenden Domänen nachweisen.
Die eukaryontischen Repressoren können auf unterschiedliche Weise
wirken. Zumeist befinden sie sich nicht in einer direkten Konkurrenz
mit der RNA-Polymerase um eine Bindungsstelle, sondern blockieren
entweder die Bindungsstelle des Aktivators, binden selbst an die
Aktivierungsdomäne des Aktivators und verhindern die Bindung der
Polymerase oder sie binden z.B. an eine der ersten Stufen der TATA-Faktoren
und blockieren so die Anlagerung der allgmeinen
Transkriptionsfaktoren.
Die Regulationsproteine der Eukaryonten bestehen häufig aus einem
Komplex von mehreren kurzen Peptiden, die nur an der DNA einen Komplex
ausbilden können, der dann die Transkription aktiviert oder hemmt.
Ein besonders auffallendes Beispiel einer solchen komlexen Kontrolle
ist das even-skipped- oder eve-Gen bei Drosophila.
Der Embryo befindet sich in einem frühen Entwicklungsstadium in einem
sogenannten Syncytium, in dem er aus einer riesigen Zelle mit mehreren
Kernen besteht. In dessen Cytoplasma sind verschiedene
Regulatorproteine ungleichmässig verteilt. Durch diese
Ungleichverteilung entsteht eine Positionsinformation. Da dadurch die
einzelnen Kerne unterschiedlichen Regulatoren ausgesetzt sind,
beginnen sie unterschiedliche Proteine zu synthetisieren.
Die regulatorischen Sequenzen des eve-Gens lesen die Konzentration der
Regulatoren, was zu einer unterschiedlichen Expression des Gens in
sieben Streigen führt. Innerhalb dieses Streifens von fünf bis sechs
Kernen breite sind diese entlang der anterior-posterioren Achse
präzise ausgerichtet.
Die Regulatorgen-Region von eve ist ca. 20.000 Nukleotide lang und
besteht aus einer Reihe einfacher Moduln. Mit Reportergenen konnte man
nachweisen, dass bestimmte Regionen für das Ausbilden bestimmter
Steifen verantworlich sind.
Das Streifen-2-Modul enthält z.B. zwei Aktivatoren (bicoid und
huchback) und zwei Repressoren (Krüppel und giant). Somit werden vier
Positionssignale integriert und nur wenn diese alle vorhanden
bzw. nicht vorhanden sind, kann die Transkription von eve starten.
Dabei ist es wichtig, dass sich die Module gegenseitig nicht
beeinflussen.
Häufig findet man bei komplexeren Kontrollmechanismen auch eine
Regulation der Regulatorproteine selbst. Dies kann durch
Phosphorylierung, durch Freigabe aus einem Komplex oder ähnliches
geschehen.
Dadurch, dass der Rand eines jeden Basenpaares aus der Helix
herausragt und dort die Bildung spezifischer Wasserstoffbrücken
ermöglicht, können Regulatorproteine die DNA lesen und an bestimmte
Sequenzen binden.
Da die Doppelhelix der DNA auch nicht absolut gleichförmig ist, kann
das Protein seine Bindungsstelle durch Interaktion mit der Oberfläche
erkennen.
Bei vielen Regulatorproteinen findet man spezifische DNA-bindende
Motive:
Das Helix-Turn-Helix-Mitv ist eines der einfachsten und häufigsten
DNA-bindenden Motive. Es besteht aus zwei 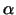 -Helices, die durch
eine gestreckte Aminosäurekette, die den Turn verursacht, verbunden
sind. -Helices, die durch
eine gestreckte Aminosäurekette, die den Turn verursacht, verbunden
sind.
Eine der Helices, die Erkennungs-Helix tritt mit der DNA in Verbindung
und ist massgeblich an dem Erkennungsprozess beteiligt. Neben diesem
Motiv treten weitere Teile ausserhalb dieser Region mit der DNA in
Kontakt und sorgen für die Feinabstimmung der Bindung.
Bei Drosophila gelang es, homöotische Selektorproteine zu
charakterisieren, die für die Entwicklung der Fruchtfliege
verantworlich sind. Alle diese Proteine enthielten eine Homöodomäne in
Form eines HTH-Motivs, das in immer die gleiche Struktur - die
Homöodomäne eingebettet ist.
Während das HTH-Motiv nur Aminosäuren verwendet, benutzt das
Zinkfingermotiv Zink als Strukturkomponente.
Man kann die Zinkfingermotive in mehrere Gruppen unterteilen.
Bei einer Gruppe bestehen die Motive aus einer 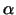 -Helix und
einem -Helix und
einem 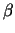 -Faltblatt, die durch ein Zinkatom zusammengehalten
werden. Die -Faltblatt, die durch ein Zinkatom zusammengehalten
werden. Die 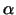 -Helix des Zinkfingers kann mit der DNA Kontakt
aufnehmen und durch mehrere hintereinander hängende Zinkfinger kann so
eins starke und spezifische Wechselwirkung mit der DNA entstehen. -Helix des Zinkfingers kann mit der DNA Kontakt
aufnehmen und durch mehrere hintereinander hängende Zinkfinger kann so
eins starke und spezifische Wechselwirkung mit der DNA entstehen.
Einen anderen Typ von Zinkfinger findet man bei intrazellulären
Rezeptorproteinen. Dessen Struktur ist der des HTH-Motivs ähnlich: Es
werden zwei 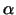 -Helices um ein Zinkatom herum gefaltet. Die
bilden - wie auch die HTH-Motiven - einen Dimer, bei der jede -Helices um ein Zinkatom herum gefaltet. Die
bilden - wie auch die HTH-Motiven - einen Dimer, bei der jede
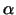 -Helix mit der grossen Furche der DNA in Kontakt treten kann. -Helix mit der grossen Furche der DNA in Kontakt treten kann.
Neben der Erkennung der grossen Furche durch helicale Elemente gibt es
auch Strukturen, bei denen die grosse Furche von zwei
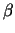 -Faltblättern erkannt wird. -Faltblättern erkannt wird.
Beim Leucin-Zipper-Motiv bilden zwei Helices eine kurze Doppelwedel,
die durch hydrophobe Interaktionen zusammengehalten wird. Die Bildung
von einem Dimer auf diese Art und Weise bietet den Vorteil, dass eine
sogenannte kombinatorische Kontrolle ausgeübt wird, bei der es möglich
ist, dass zwei unterschiedliche Monomere zusammen einen Heterodimer
bilden, der eine andere Sequenzspezifität als die Homodimere
aufweist.
Ein anderes DNA-bindendes Motiv ist das Helix-Loop-Helix-Motiv
(HLH). Bei diesem Motiv ist eine kurze 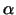 -Helix mit einer
weiteren durch eine kurze Schlaufe verbunden. -Helix mit einer
weiteren durch eine kurze Schlaufe verbunden.
Diese Motive bilden ähnlich den Leucin-Zipper-Motiven Dimere, bei
denen sich die beíden langen Helices in zwei benachbarte Furchen
lagern. durch die Bildung von Heterodimeren kann es wie oben zu neuer
Sequenzspezigität kommen oder - im Fall von einer Dimerisierung mit
einem HLH-Protein, dessen lange Helix nicht in der Lage ist, mit der
DNA Kontakt aufzunehmen - zu einer Inaktivierung des HLH-Mechanismus
kommen.
Bei einem gel mobility-shift assay verwendet man eine radioaktiv
markierte DNA, von der man animmt, dass sie von einem bestimmten
Protein gebunden wird.
Bei der Affinitätschromatorgraphie kann synthetisiert man ein kurzes
Nukleotid, zu dem man die an dieser Stelle bindenden Proteine
isolieren möchte. Diesesen Oligo bindet man fest an eine Martix aus
z.B. Agarose und stellt damit eine Säule her. Über diese lässt man
dann einen Zellextrakt laufen und eluiert aus der Säule dann die
Proteine, die fest an die Nukleotide gebunden haben.
Methylierung
Neben der Kontrolle der Genexpression durch Proteine ist die
Methylierung ein wichtiger Mechanismus, um die Genexpression zu
regulieren. Bei den Säugern wird nur eine C-G-Gruppe methyliert - und
auch nur dann, wenn sie von einer G-C-Gruppe gefolgt ist. So kann die
Methylierung einer DNA auf die Tochterstränge übertragen und in
mehreren Generationen von Zellen unverändert vererbt werden.
Dieses System lässt sich mit Enzymen aus Bakterien, die durch
Methylierung ihre eigene DNA vor ihren Restriktionsenzymen schützen,
leicht untersuchen.
Man nimmt an, dass in der Eizelle eine de novo-Methylierung
stattfindet und später die so erzeugten Muster nur noch dann ergänzt
werden, wenn sich die DNA geteilt hat und die Tochterstränge
methyliert werden müssen.
Man vermutet, dass die Methylierung nicht direkt zu einer
Inaktivierung führt, sondern dass die methylierten Bereiche bestimmte
Proteine binden, die dann wiederum zu einer Inaktivierung des Gens
führen.
Die Methylierung kann dazu führen, dass ein methyliertes C zu durch
Desaminierung zu einem T wird und dieses von den Reparaturmechanismen
nicht erkannt wird. Auf diese Weise sind viele der CG-Paarungen im
laufe der Evolution der Wirbeltiere verschwunden. Eine Ausnahme bilden
die sogenannten CG-Inseln. Diese findet man in den Promotorregionen
der Haushaltsgene und in den Bereichen von gewebespezifischen Genen.
Man führt dies darauf zurück, dass diese Bereiche schon in der
Keimbahn unmethyliert vorlagen und deshalb von dem oben genannte
Effekt verschont blieben.
Da diese Inseln häufig den Beginn einer Transkriptionseinheit
darstellen, kann man sie benutzen, um neue Gene zu klonieren.
Bei den diploiden Zellen der Säuger ist die Aktivität eines Gens in
einigen Zellen davon abhängig, ob die Zelle dieses vom Vater oder der
Mutter geerbt hat.
Dies kann man z.B. bei Mäusen zeigen, bei denen eines der IGF-2-Gene
inaktiviert wird. Wenn das Gen der Mutter inaktiveriert ist,
entwickeln sich die Mäuse normal. Wird hingegen das Gen des Vaters
inaktiviert, führt dies zu einem veränderten Phänotyp.
Man vermutet, dass dieser genetische Stempel durch die Methylierung
hervorgerufen wird.
|





