Unterabschnitte
Transkription
Die RNA-Polymerase entwindet an ihrem Vorderende die DNA und lässt so
im Inneren die Bildung von DNA-RNA-Hybriden zu. Die Ausdehnung dieser
Transkriptionsblase beträgt ca. 12 bis 20 Basenpaare. Die Stelle, an
der die RNA mit der DNa hybridisiert ist auf eine Länge von 3 bp
beschänkt. Die meisten RNA-Polymerasen haben an ihrere Oberfläche eine
Furche, in der die DNA zu liegen kommt.
Die RNA-Polymerase kontrolliert das passgenaue Binden des nächsten
Nukleotids. Sehr wahrscheinlich werden die falschen Nukleotide von der
Polymerase von der Bindung abgehalten.
Die Bewegung der RNA-Polymerase ist in einigen Bereichen gleichförmig
und in anderen hingegen ruckartig. Wird sie in ihrer Synthese
angehalten (weil z.B. ein Nukleotid fehlt), spaltet sie unter Katalyse
von zwei Faktoren GreA und GreB das soebensynthetisierte 3'Ende der
RNA ab und bewegt sich ein Stück weit an der RNA zurück, um die
Synthese neu zu beginnen.
Die Transkription lässt sich in mehrere Phasen einteilen:
Bie Erkennung der Matrize durch die RNA-Polymerase bildet den ersten
Schritt, durch den die Transkriptionsblase entsteht. Darauf folgt die
Initiation, in der die ersten Nukleotide binden. Diese Phase ist dann
beendet, wenn sich die Polymerase vom Promotor entfernt hat und geht
dann in die Elongation über. Bei diesem Prozess wandert die Blase und
verlängert das Transkript, bis eine Terminator-DNA-Sequenz erreicht
ist und die Termination stattfindet.
Per Definition ist die RNA-Polymerase die kleinste Einheit des
Tranksriptionskomplexes, die noch in der Lage ist, selbstständig RNA
zu synthetisieren.
Die am besten charakterisierte Polmerase ist die von E. coli. Diese
Polymerase ist ein Holoenzym, das sich aus mehreren Untereinheiten
aufbaut:
Die 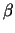 -und die -und die 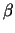 '-Untereinheit bilden das katalytische
Zentrum. Zwei '-Untereinheit bilden das katalytische
Zentrum. Zwei 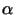 -Untereinheiten kontrollieren den Zusammenbau
des Kernkomplexes der Polymerase. Die -Untereinheiten kontrollieren den Zusammenbau
des Kernkomplexes der Polymerase. Die 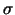 -Untereinheit
schliesslich ist dafür verantwortlich, dass die Polymerase einen
spezifischen Promotor erkennt. Dieser Komplexe Aufbau beruht darauf,
dass das Enzym in der Lage sein muss, mit anderen Faktoren, die die
Transkription regulieren, zu interagieren. -Untereinheit
schliesslich ist dafür verantwortlich, dass die Polymerase einen
spezifischen Promotor erkennt. Dieser Komplexe Aufbau beruht darauf,
dass das Enzym in der Lage sein muss, mit anderen Faktoren, die die
Transkription regulieren, zu interagieren.
Der 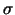 -Faktor wird freigesetzt, sobald die RNA acht bis neuen
Basen lang ist. Dadurch veränder sich die Form der RNA-Polymerase. Sie
wird kürzer und bedeckt einen kleineren DNA-Abschnitt. Dadurch geht
sie von der Initiation in die Elongation über. Der Sigma-Faktor
gewährleistet, dass die RNA-Polyermase nur an den Promotoren stabil
bindet. Das restliche ,,Core``-Enzym ist nicht spezifisch in
seiner Bindung, sonden wird allein durch den -Faktor wird freigesetzt, sobald die RNA acht bis neuen
Basen lang ist. Dadurch veränder sich die Form der RNA-Polymerase. Sie
wird kürzer und bedeckt einen kleineren DNA-Abschnitt. Dadurch geht
sie von der Initiation in die Elongation über. Der Sigma-Faktor
gewährleistet, dass die RNA-Polyermase nur an den Promotoren stabil
bindet. Das restliche ,,Core``-Enzym ist nicht spezifisch in
seiner Bindung, sonden wird allein durch den 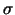 -Faktor
gesteuert. -Faktor
gesteuert.
Das Auffinden der Bindungsstellen durch die Polymerase erfolgt im
Allgemeinen zu schnell, als dass es auf einer einfachen Diffusion
beruhen kann. Eine Theroe besagt, dass die Polymerase nach ihrer
Bindung an das Genom die gebundene Sequenz so lange austauscht, bis
ein Promotor erreicht ist. Eine alternative Theorie postuliert eine
Bewegung der Polymerase entlang der DNA, bis sie eine Bindungsstelle
erreicht.
Die Bindung des Core-Enzyms an die DNA ist in Abwesenheit des
Sigma-Faktors immer nur eine lockere Verbindung.
Der Sigma-Faktor bindet spezifisch an die Promotoren. Dies sind
Sequenzen, die von Proteinen erkannt werden. Die Grösse dieser
Strukturen nimmt mit der Grösse des Genoms zu, da immer ausgeschlossen
werden muss, dass sich die Promotor-Sequenz an einer anderen Stelle
zufällig im Genom befindet.
Bei E. coli scheint die genaue Sequenz eines Promotors nicht wichtig
zu sein. Einige, in den meisten Promotoren hochkonservierte, Sequenzen
bezeichnet man als Consensussequenzen. Man findet Consensussequenzen
am Beginn des Promotors (häufig CAT), in der Region von -10 bp (meist
TATAAT) und eine weitere konservierte Sequenz bei -35 bp (meist
TTGACA).
Eine Mutation kann einen Promotor abschwächen (down-Mutation) oder
dessen Funktion verstärken (up-Mutation). Häufig aber nicht immer ist
eine up-Mutation dadurch gekennzeichnet, dass sich die Sequenz in
ihrer Hologie näher an die Sequenz der Consensussequenzen annähert.
Normalerweise quantifiziert man die Mutation durch einen Bindungstest
der Polymerase in vitro.
Die Bindung der Polymerase kann durch das sogenannte Footprinting
charakterisiert werden. Bei diesem Verfahren wird die DNA mit einer
Endonuklease partiell abgebaut. Der Bereich, in dem die Polymerase
gebunden hat, ist vor dem Aufbrechen der Phosphodiesterverbindungen
geschützt.
Isoliert man dann die geschnittenen Stücke, welche an ihrem Ende
radioaktiv markiert sind, erhält man ein dichtes Muster von Banden,
bei denen nur die Stelle, an die Polymerase gebunden hatte, leer
bleibt und einen ,,Fussabdrck`` aufweist.
Durch eine (negative) Überspiralisierung wird die Transkription
gefördert. Das Zwillingsdomänenmodell besagt, dass die RNA-Polymerase
vor sich in der DNA eine positive Überspiralisierung erzeugt, während
es hinter sich eine negative Überspiralisierung zurücklässt.
Um diese Veränderungen auszugleichen werden die Enzyme Topoisomerase,
die die DNA entspannt und Gyrase, die eine Spannung einführt,
benötigt.
Die Initiation kann durch unterschiedliche Sigma-Faktoren kontrolliert
werden. Die Sigma-Faktoren werden vor allem dann ausgetauscht, wenn
die Transkription sich von Grund auf verändert. Grund für eine solche
Umorganisation kann ein z.B, ein Hitzeschock sein.
Bei einem Temperaturanstieg verändert sich die Zusammensetzung der
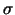 -Faktoren und führt dazu, dass andere Proteine hergestellt
werden. -Faktoren und führt dazu, dass andere Proteine hergestellt
werden.
Durch Unterschiede in den Konsensussequenzen kann durch
unterschiedliche 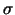 -Faktoren ein unterschiedliches Genset ein-
oder ausgeschaltet werden. Die Sigma-Faktoren nehmen mit der Sequenz
im Bereich -35 und -10 Kontakt auf. -Faktoren ein unterschiedliches Genset ein-
oder ausgeschaltet werden. Die Sigma-Faktoren nehmen mit der Sequenz
im Bereich -35 und -10 Kontakt auf.
Neben dem Hitzschock können auch andere Umwelteinflüsse wie eine
Infektion durch einen Phagen oder der Eintritt in die Sporulation die
Zusammensetzung der 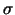 -Faktoren in der Zelle verändern. -Faktoren in der Zelle verändern.
Bei einer Phageninfektion werden die frühen Gene des Phagen von der
Polymerase des Wirtes umgesetzt. Unter den so umgesetzten Genen
befindet sich auch ein neuer Sigma-Faktor, der den bakteriellen Faktor
ersetzt. Die dadurch umgesetzen mittleren Gene sind dafür
verantwortlich, dass die Polymerase nach ihrer Bindung nur noch die
späten Gene umsetzen kann.
Bei der Sporulation ist der Nahrungsmangel ein entscheidender
auslösender Faktor. Dadurch führt die Mutterzelle eine Mitose durch
und kapselt eine Tocherzelle als Spore ab, während die andere Zelle
lysiert wird. In der sporulierenden Zell werden unterschidliche
Polymerase-Arten aktiv. Sie enthalten das gleiche Core-Enzym,
allerdings einen anderen 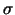 -Faktor. Dieser wird durch die
Kontrolle von phosphorylierten Sporulationsfaktoren von dem normalen -Faktor. Dieser wird durch die
Kontrolle von phosphorylierten Sporulationsfaktoren von dem normalen
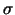 -Faktor synthetisiert. -Faktor synthetisiert.
Die Transkription wird an einer bestimmte Terminationssequenz
beendet. An einigen Termination können Antiterminatoren durch Bindung
und Interaktion mit der Polymerase die Termination verhindern, was zu
einem Überlesen der Stelle führt. Die Termination ist von dern bereits
transkribierten - nicht aber von den noch zu transkribierenden
Abschnitten abhängig.
An einigen Stellen kann dasn Core-Enzym in vitro die Transkription
ohne die Hilfe von anderen Faktoren beenden. Diese Stellen werden
intrinsische Terminatoren genannt. Im Gegensatz dazu wird die
Transkription an den Rho-abhängigen Terminatoren nur in Anwensenheit
eines Rho-Faktors beendet.
Die intrinsischen Terminatoren bilden eine auf eine UUUU-Sequenz
folgende Haarnadelstruktur aus, die sehr wahrscheinlich die Polymerase
zu einer relativ langen Pause veranlasst, so dass diese dann
terminiert. Wenn sie Polymerase anhält, kann sich die PolyAU-Sequenz
lösen, da die Bindung zwischen A und U relativ schwach ist.
Der Faktor Rho ist nur an der Termination der Transkription
beteiligt. Dieser Faktor bindet an die RNA und ,,verfolgt``
die RNA-Polymerase auf dem Strang. Wenn die Polymerase an einer
Terminationssequenz anhält, dann erreicht Rho die Polymerase und
verursacht unter ATP-Hydrolyse die Trennung des RNA-DNA-Hybrids und
beendet so die Transkription.
Normalerweise verhinder die Translation und die Bindung der Ribosomen
die Aktivität von Rho. Bei einem als Polarität bezeichneten Phänomen
verhindert eine Mutation in einem Gen die Transkription weiterer Gene
innerhalb der Transkriptionseinheit. Die könnte darin begründet sein,
dass sich das Ribosom durch die Mutation ablöst und Rho so in der Lage
ist, die Transkription vorzeitig zu beenden.
Bei einigen Phagen und auch in Regelkreisen von Bakterien ist die
Antitermination ein wichtiger Faktor. Bei den Phagen befindet sich
zwischen den frühen und den späten Genen eine
Terminatinossequenz. Nach der Aktivierung der frühen Gene werden
Faktoren produziert, die eine Antitermination an dieser Stelle
bewirken. Das Antiterminationsprotein gestattet es der Polymerase, die
Terminationssequenz zu überlesen.
Die Faktoren binden vermutlich dann an die RNA-Polymerase, wenn sie
eine bestimmte Sequenz, die nut-Sequenz erreicht. Dieser Faktor (pN)
bindet an das Core-Enzym ähnlich dem Sigma-Faktor und verhindert, dass
die Polymerase an einer Terminationssequenz stoppt. So kann diese
nicht von Rho eingeholt werden. Ein anderer Faktor pQ wirkt auch eine
andere Weise, indem er die Polymerase so modifiziert, dass sie an
keinem Terminator mehr hält.
Ein Gen, das die Transkription eines anderen Gens kontrolliert wird
Regulatorgen; das kontrollierte Gen wird Strukturgen genannt.
Ein Repressorprotein, das die Transkription eines Gens verhindert,
bindet an dem in der Nähe des Promotors liegenden Operator und hindert
so die Polmerase an einer Bindung an die DNA.
Als klassisches Beispiel für das Operon-Modell gelten die Strukturgene
lacZYA, die der Zelle die Aufnahme und den Abbau von Laktose
ermöglichen.
Die Einheit aus diesen Struktur- und ihren Regulationgenen wird als
Operon bezeichnet. Die Gene werden durch negative Regulation
kontrolliert, d.h. sie werden transkribiert, bis das Regulatorprotein
sie abschaltet.
Das Vorhandensein von Laktose induziert eine Transkription der
entsprechenden Gene. Im umgekehtre Fall, wenn das Produkt eines Gens
dessen Synthese hemmt, dann spricht man von Repression.
Der Co-Repressor oder der Induktor wirkt über eine entsprechendes Protein
auf die DNA ein. Die Bindung an dem Protein (dem Repressor) bewirkt
über eine allosterische Kontolle dessen Bindung, was zu einer
koordinierten Regulation der Synthese führt.
Wenn der Repressor den Induktor nicht binden kann, ist dies eine dem
Wildtyp gegenüber dominante Mutation, die den Repressor fest mit der
DNA verbindet. Diese Mutation wird auch als dominant-negativ
bezeichnet, da die ,,schlechten`` Untereinheiten der Mutatante
auch an dem anderen Allel eine Induktion verhindern.
Der Operator, die Erkennungsstelle des Repressors ist ein Palindrom
und spiegelt den symmetrischen Aufbau des Proteins wieder. Die Wirkung
des Induktors wird durch unterschiedliche Theorien zu erklären
versucht. Nach einer Theorie befindet sich der Repressor in einem
Gleichgewicht zwischen DNA-gebundener und freier Form, das durch die
Bindung des Induktors zu Gunsten der freien Form verschoben wird. In
einem anderen Modell ist der Repressor fest an die DNA gebunden und
löst sich erst durch die Bindung des Indukors.
Richtig ist sehr wahrscheinlich das letztere Modell.
Der Repressor bindet die DNA über ein Helix-Turn-Helix-Motiv. Dieses
Motiv bildet zusammen mit einerm Schanier als Verbindung zum
Kernprotein das Kopfstück des Proteins. Wenn der Induktor an das
Kernstück bindet, verändert sich dessen Konformation so, dass die
beiden Köpfe des Repressor-Dimers auseinander bewegen und die Bindung
an die DNA verloren geht.
Für eine vollständige Repression ist das gleichzeitige Binden von zwei
zu einem Tetramer vereinigten Dimeren an zwei aufeinanderfolgenden
Operatoten notwendig. So kann die Transkription stufenweise geregelt
werden.
Der Repressor verstärkt die Bindung der Polymerase und hinder sie so
daran, einen Initiationskomplex auszuformen. So kann nach dem Binden
des Induktors die Polymerase direkt mit der Transkription beginnen.
Man kann über Messungen an in vitro Systemen eine
Gleichgewichtskonstate für die Bindung und Dissoziation des Repressors
erstellen. Man kann daraus errechenen, dass 0,01% aller
Repressorproteine in einer ungebundenen Form vorliegen. Die Fähigkeit
eines Proteins seine Zielstelle zu binden ist von der Grösse des
Genoms, der Spezifität des Proteins, der Proteinmenge und der Relation
zwischen Anzahl der Proteine und Anzahl der Bindungsstellen ab.
Wenn der Induktor bindet, sinkt die Affinität des Repressors zur DNA
und er löst sich leichter. Er bindet dann - wie die nicht an den
Operator gebundenen Repressoren es im Normalfall tun - an eine
beliebige DNA-Sequenz mit niedriger Affinität.
Das Auffinden des Operators geschieht sehr wahrscheinlich nach der
gleichen Methode, wie auch die Polmerase ihre Sequenz findet: der
Repressor bindet unspezifisch und bewegt sich dann an der DNA entlang
bis er den Operator findet. Im Normalzustand ist das Niveau des
Respressors so geregelt, dass noch einige Gene abgelesen werden
können; allerdings bedeutet dies auch, dass bei Vorhandensein des
Induktors keine Bindung des Repressors erfolgt. Bei einer höheren
Konzentration würde der Repressor auch bei vorhandenem Induktor noch
einen grossen Anteil der Operatoren binden.
Im Gegensatz zum lac-Repressore kontrollieren einige andere
Repressoren mehrere vertreut liegende Gene gleichzeitig. Dazu muss
jeder Wirkort die gleiche DNA-Bindungssequenz aufweisen.
Grundsätzlich kann man bei einem Operon-Modell zwischen einer
negativen Kontolle, bei der die Gene exprimiert werden, wenn sie nicht
durch einen Repressor abgeschaltet werden und einer positiven
Kontrolle, bei der es eines Aktivators bedarf, damit die Gene
transkribiert werden. Auch der Austausch eines Sigma-Faktors ist eine
positve Kontrolle.
In Abhängigkeit von einer niedermolekularen Substanz unterscheidet man
induzierbare Operons, die in Anwesenheit der Substanz aktiv und
reprimierbare sind nur bei Abwesenheit der Substanz aktiv.
Beides kann über positive und negative Kontrolle erreicht werden.
Eine Art der postiven Regulation findet man an Promotoren, an denen
die Polymerase nur in Anwesenheit eines weiteren Faktors binden und
die Transkription starten kann.
Eine solche Regulation findet man bei dem Mechanismus der
Katabolitrepression, bei der die Expression mehrere Operons für
Lactose, Galactose, etc. zu Gunsten der Glukose verhindert wird.
Glukose reduziert dazu die Konzentration von cAMP in der Zelle. cAMP
bindet normalerweise an ein CAP-Protein, das für die Transkription
mehrerer Gene erforderlich ist. Durch ein Absinken des cAMP-Spiegels
kann CAP nicht mehr binden und die Polymerase kann die Transkription
nicht mehr initiieren.
CAP wirkt, indem die von ihm kontrollierten Polymerasen eine schlechte
Bindung an die -10 und die -35 - Sequenz haben. Die CAP interagiert
mit der Polymerase und der DNA und bewirkt so, dass eine Bindung zu
stande kommt. Man vermutet, dass CAP an der gleichen Seite wie die
Polymerase bindet und mit deren 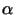 -Einheit interagiert. -Einheit interagiert.
Wenn ein Bakterium nicht genügend Nährstoffe zum Wachstum vorfindet,
stellt es einen grossen Teil seiner normalen Aktivität ein. Dies
bezeichnet man als stringente Kontrolle. Die Synthese der rRNA und der
tRNA wird verringert und senkt dadurch die gesamte RNA-Synthese auf
fünf bis zehn Prozent.
Durch die stringente Kontrolle häufen sich zwei ungewöhnliche
Nucleotide an: ppGpp und pppGpp sind Guaninphosphaten, bei denen
sowohl die 5'- als auch die 3'-Postition mit Phosphaten versehen ist.
Diese Nukleotide kontrollieren eine grosse Zahl an Zellaktivitäten und
senkt die Vermehrungsgeschwindigkeit.
Der ganze Mechanismus wird dadurch, dass eine unbeladene tRNA an der
Bindungsstelle des Chromosoms bindet aktiviert. Dies löst eine
Leelaufreaktion aus, welche über den Stringenzfaktor die Konzentration
der (p)ppGpp kontrolliert. Die eigentliche Synthese geschieht direkt
an den Ribosomen.
Die Synthese geschieht dadurch, dass der Stringenzfaktor RelA eine
Phosphatgruppe vom ATP auf das Guaninnukleotid überträgt. Sobald sich
die Gegebenheiten wieder normalisieren, wird das (p)ppGpp wieder
abgebaut.
Neben der Kontrolle der Transkrition kann ein Operon auch die
Translation kontrollieren wenn es für Bestandteile des
Proteinsyntheseapparates codiert.
Dabei wirkt ein Protein als Repressor, das an der mRNA in der
Erkennungsregion bindet und so die Aktivität der Ribosomen
verhindert.
Ein anderer Mechanismus besteht darin, dass das zweite Cistron so
lange blockiert ist, bis diese Blockade durch das Genprodukt des
ersten Cistrons aufgelöst wurde.
Diese Kontrollmechanismen gewährleisten, dass die Proteine immer in
dem Ausmass synthetisiert werden, wie es die Umweltbedingungen
erlauben.
Von einer autogenen Regulation spricht man, wenn ein Protein seine
eigene Synthese (in dem Fall durch Regulation der Transkription)
regelt. Diesen Mechanismus findet man bei Histonen und den Proteinen
der ribosomalen Untereinheiten. Die Voraussetzung ist, dass das
Protein eine Bindungsstelle für seine DNA-Sequenz besitzt. Bei den
ribosomalen Proteinen ist die z.B. von der Konzentration der rRNA
abhängig. Wenn diese in einer ausreichend hohen Konzentration
vorliegt, dann binden die Proteine an die rRNA; wenn deren
Konzentration unter ein kritisches Limit fällt, verhindern sie ihere
eigene Translation.
Dieser Mechanismus ist weit verbreitet und bei einem mutierten Gen
häufig defekt. Bei einem System, das sich selbst begrenzt, spricht man
von einer intrinsischen Kontrolle; bei einer Begrenzung von aussen her
von einer extrinsischen Kontrolle.
Mehrere Operons werden durch Attnuation geregelt. Die Attenuation
beeinflusst die Fähigkeit der Polymerase über einen intrinsischen
Terminator, den Attenuator am Beginn der Sequenz hinweg zu lesen.
Der Attenuator besteht aus einer Haarnadelstruktur, die sich auf ein
äusseres Signal hin bildet. Der Mechanismus ist abhängig von der
Translation der RNA durch das Ribosom. Ist diese bereits weiter
fortgeschritten, beendet dies die Transkription.
Ein solcher Attenuationsmechanismus verstärkt die Wirkung des
Tryptophanrepressors, indem es in Anhängigkeit von der
Tryptophankonzentration die Transkription unterbricht. Hier ist der
Mechanismus sehr wahrscheinlich so, dass die Ribosomen, die die RNA
der Leader-Sequenz
translatieren bei einem Tryptophanmangel inne halten und so die
Transkription erlauben und sie andondten unterbinden.
In einigen Fällen findet man auch Regulationsmechanismen, die nicht
auf Proteinen, sondern auf RNA beruhen. Für die Wirkungsweise ist ein
allosterischer Mechanismus ebenso denkbar wie eine direkte Interaktion
mit der Zielsequenz. In der Praxis findet man allerdings keine
allosterischen Effekte, sondern nur eine direkte Interaktion durch
Antisense-RNA, die dann entweder die Bindungsstelle der Ribosomen
blockiert, die Stabilität beeinflusst, da sie einen Doppelstrang als
Signal für Exonukleasen bildet oder sie bildet eine Art
Terminationssignal, das die Transkription vorzeitig beendet.
Einen ungewöhnlichen Mechanismus findet man bei dem Phagen T7. Ein
polycistronisches Gen wird in eine RNA umgschrieben, die Anschliessend
durch die RNase III in mehrere RNAs der einzelnen Proteine gespalten
wird.
Das Schittsignal für diese RNase ist eine von der RNA ausgebildete
Doppel-Helixstruktur.
Bei Säugern findet man ein ähnliches Prozessing bei den ribosomalen
Untereinheiten. Die 45S-Untereinheit enhält die 18S, die 5,8S und die
28S-Untereinheit und ist stark methyliert. Sehr wahrscheinlich
markieren die Methylgruppen die Bereiche, die später zur RNA werden.
Die rRNAs der Bakterien werden auf einem ähnlichen Weg aus einer
Vorläufer-RNA durch RNase III freigesetzt.
Kontrolle der Transkription bei Eukaryonten
Im Zellkern der Eukaryonten sind drei Polymerasen für die
Transkription verantworlich: Die Polymerase I transkribiert die rRNA,
die Polymerase II die mRNA und die Polymerase III die tRNA und andere
kleine RNAs.
Bei allen Transkriptionen spielen Faktoren, die an die DNA binden eine
wichtige Rolle. Bei der Polymerase II arbeiten drei Hauptgruppen an
der Initiation der Transktiption:
- Allgemeine Transkriptionsfaktoren sind das Grundgerüst
für die Transkription, bilden mit der RNA-Polymerase einen Komplex
und sind für jede Transkription notwendig.
- upstream-Faktoren binden stromaufwärts an die DNA,
wobei sie an jedem Promotor binden und nicht reguliert werden.
- Induzierbare Faktoren binden an Response-Elemente auf
ähnliche Art wie die upstream-Faktoren, nur dass diese Gruppe
reguliert wird.
Ein Abschnitt der DNA, der auf die Transkription einwirkt, sich aber
in einiger Entfernung von dem Promotor befindet, wird Enhancer
genannt.
Im Allgemeinen ist die Regulation bei den Eukaryonten positive; eine
Repression findet man selten.
Die Polymerase I ist im Nucleolus loakalisiert; die anderen beiden im
Nucleoplasma. Um die drei Polymerasen zu unterscheiden kann man ihre
unterschiedliche Empfindlichkeit gegenüber 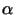 -Amanitin
ausnutzen. Die Polymerase II reagiert darauf sehr empfindlich; die
POlymerase I gar nicht und die Reaktion der Polymerase III schwankt
zwischen den Spezies. -Amanitin
ausnutzen. Die Polymerase II reagiert darauf sehr empfindlich; die
POlymerase I gar nicht und die Reaktion der Polymerase III schwankt
zwischen den Spezies.
Die Polymerasen sind alle aus mehreren Untereinheiten aufgebaut. Bei
der Polymerase II der Hefe fand man 10 oder 11 Untereinheiten.
Ein wichtiges Merkmal der Polymerase II ist eine Ansammlung von kurzen
repeats am C-Terminus. Werden diese deletiert, ist dies meist lethal.
Die RNA-Polymerase ähnelt der der Bakterien.
Die Untersuchungen stützen sich auf Versuche mit Xenopus-Oocyten,
Transfektionssysteme, Transgene Systeme und in vitro-Systeme.
Auf diese Weise versucht man, einen Promotor zu charakterisieren,
indem man die DNA verändert und dann in dem Testsytem
untersucht. Durch sukzessives Abschneiden eine Endes kreist man die
Promotor-Sequenz immer weiter ein. Hat man die Grenzen eines Promotors
ermittelt, versucht man durch Punktmutationen die relevanten Basen zu
identifizieren. Entweder lässt sich - wenn das entsprechende Protein
an mehrere Sequenzen bindet - eine Consensussequenz ermitteln oder
man analysiert die Bindung eines Proteins durch Footprinting.
Bei der Polymerase I findet man nur einen einzigen Typ von Promotor,
der die Transkription der rRNA steuert, aus der hinterher die
einzelnen rRNAs - mit Ausnahme der 5S-rRNA, die von der Polymerase II
transkribiert wird - gespleisst werden.
Der Core-Promotor liegt im Bereich von -45 bis +20 und wird durch ein
upstram control element bei -180 bis -107 verstärkt. Die beiden
Sequenzen stimmen in weiten Teilen miteinander überein. Dort steuern
zwei Hilfsproteine die Transkription. UBF bindet spezifisch an die
beiden Sequenzen. Durch eine Bindung kann auch SL1 binden und an
diesen Komplex lagert sich dann die Polymerase I an. SL1 ist für die
sequenzspezifische Erkennung notwendig.
Die Promotoren der 5S-rRNA und der tRNA befinden sich stromabwärts
innerhalb der Sequenz, während z.B. die snRNA ihren Regulator
stromaufwärts besitzt.
Bei der 5S-rRNA befindet sich der Promotor zwischen +55 und +80. Die
Wirkung der internen Promotoren beruht darauf, dass zunächst
sogenannte assembly Faktoren an die DNA binden, wobei einer in der
Region des Promotors, der andere in Richtung des Startpunktes
bindet. An den letzteren kann dann wiederum der eigentliche
Initationsfaktor binden, der mit der Polymerase interagiert und so die
Transkription initiiert.
Bei den stromaufwärts gelegenen Promotoren sind den Promotoren der
Polymerase II sehr ähnlich und kann an dem upstream des Starts
gelegenen TATA-Element geschehen, wird allerdings durch die weiter
upstream liegenden Sequenzen PSE und OCT beeinflusst. An dem
TATA-Element binden Proteine, die die RNA-Polymerase III an den
richtigen Startpunkt bringen.
Bei allen an der Polymerase III wirksamen Faktoren findet man einen
Präinitiationskomplex, d.h. die Faktoren binden vor der Polymerase und
vermitteln so deren Bindung.
Die Polymerase II ist in jedem Fall auf Hilfsfaktoren angewiesen und
kann die Transkription alleine nicht initiieren.
Die dazu benötigten Faktoren werden als TFII bezeichnet. Die
Startsequenz selber ist meist ein CA, das von Pyrimidienen flankiert
ist: Py2CAPy5. Dieser Bereich wird Initiator genannt.
Der Promor selbst liegt meist in der Form einer TATA-Box vor.und liegt
25 bp upstream. Sie ist AT-reich und ähnelt der -10-Sequenz der
Bakterien. Nur wenige TATA-lose Promotoren besitzen keine
TATA-Box. Die TATA-Sequenz wird durch das TATA-binde-Protein TBP
erkannt. es bindet zusammen mit unterschiedlichen TBP-assoziierten
Faktoren an die DNA. Dieses TBP findet sich bei allen drei Polymerasen
wieder, ist jedoch mit unterschiedlichen Faktoren assoziiert und wirkt
deshalb unterschiedlich. TBP bindet die kleine Furche der DNA und
vermittelt den Kontakt und die Bindung der Polymerase. Dadruch dass
TBP die DNA um ca. 80 ,,knickt`` können die Polymerase und
die TFIIs besser binden.
Für die eigentliche Initiation reicht das TBP jedoch nicht ausM es ist
vielmehr notwendig, dass TFIIs in geordneter Reihenfolge nacheinander
an den Komplex binden. Da auch stronabwärts Faktoren binden, kann die
Transkription noch nicht beginnen. Erst nach dem der C-Terminale Teil
der Polymerase phosphoryliert wurde, kann sie sich von den TFIIs lösen
und beginnt die Transkription.
Bei den TATA-losen Promotoren ist der verlauf ähnlich, nur dient hier
der Initiator als Positionierungselement.
Da die meisten Fakrtoren aus mehreren Untereinheiten bestehen, wirken
etwa 20 Polypeptide an der Transkription mit. Die Funktion dieser
Faktoren ist dem Sigma-Faktor der Bakterien analog.
Neben der TATA-Box liegen weitere upstream gelegene Faktoren eine
entscheidende Rolle für die Initiation der Transkription. Diese binden
induzierbare Faktoren. Zwar ist die TATA-Box für den Beginn der
Transkription an einer bestimmten Stelle wichtig, allerdings sind die
upstream geglegenen Element wichtiger für die Initiierung der
Transkription.
Bei -75 findet man die CAAT-Box und bei -90 die GC-Box, die beide
einen grossen Einfluss auf den Initiatinsprozess haben. Keines der
verschiedenen Elemente ist unbedingt erforderlich; alle Elemente
trangen zur Promotorfunktion bei, können sich aber auch gegenseitig
ersetzen.
Beide Sequenzen werden von upstream-Faktoren erkannt, die - im
Gegensatz zu den induzierbaren Faktoren - konstitutiv vorhandne
sind. Man kann zeigen, dass der gesamte upstream-Bereich von Faktoren
bedeckt ist, interagieren miteinander und erreichen so eine sehr hohe
Flexibilität, die sich darin bemerkbar macht, dass man
unterschiedliche Promotor-Sequenzen nahezu beliebig mischen kann.
Es können unterschiedliche Proteine an diese Sequenzen binden und
somit ist die Expression stark vom Kontext abhängig.
Bei der Untersuchung in vitro gilt zu beachten, dass die DNA hier -
im Gegensatz zum Zellkern - nicht verpackt ist und somit auch die
Bindungsmuster der Proteine unterschiedlich sein können. Es kann sein,
dass ein Protein an den Promotor binde, diesen allerdings nicht
aktiviert. so kann das Promotorelement gleichzeitig ein Aktivator und
ein Repressor sein.
Im Allgemeinen werden die transkribierten Gene bevorzugt
repariert.
Wenn die Polymerase auf eine defekte Stelle des Matritzenstrangs
trifft, hält sie an und ermöglicht so einem anderen Protein, an sie zu
binden. Dieses Protein verdrängt dann die Polymerase und leitet die
Excisionsreparatur ein.
Die Position des Enhancers zum Promotor ist nicht festgelegt und er
kann in beiden Orientierung wirken. Ein Enhancer kann jeden Promotor
in seinem Einflussbereich stimulieren, wobei er up- wie downstream
wirken kann.
Die Enhancer liegen meist in einer wenig gepackten Form vor und weisen
viele Proteinbindungsstellen auf. Häufig findet man
Tandem-Wiederholungen und auch innerhalb eine Enhancers findet man
häufig Sequenzen mit einer unterschiedlichen Sequenz aber einer
identischen Funktion. So wird der Enhancer erst inaktiviert, wenn
weite Bereiche der Sequenz mutiert sind.
Teilweise beeinflussen die Enhancer die Transkription, in einigen
Fällen sind sie sogar deren Auslöser.
Man kann zeigen, dass die Eigenschaften des Enhancers auf der
räumlichen Nähe beruhen (lineare DNA mit einem Promotor am einen und
einem Enhancer am anderen Ende wird verstärkt transkribiert, wenn man
die Enden über eine Proteinbrücke verbindet). Man nimmt an, dass die
zwischen Promotor und Enhancer liegende Struktur eine Schlaufe bildet
und der Enhancer so - auf noch unklare Weise - seine Wirkung
entfalten kann.
Man konnte bei GAL4 zeigen, dass die Transkriptionsfaktoren für die
Bindung an die DNA und die Aktivation der Transkription
unterschiedliche Domänen besitzen. Die DNA-bindende Domäne hat dabei
in erster Linie die Aufgabe, die Aktivatorregion räumlich in die Nähe
des Promotors zu bringen.
Diese Transkriptionsfaktoren beeinflussen die Transkription über eine
Interaktion mit dem basalsen Appparat (Pol-II und TFII).
Dabei kommt es bei einige Proteinen zu einem direkten Kontakt zwischen
der Aktivatorregion und dem Transkriptionskomplex, in anderen Fällen
wird die Interaktion über Cofaktoren vermittelt.
Viele upstream- und Enhancer-Faktoren bestimmen auf diese Weise die
Geschwindigkeit des Zusammenbaus des basalen Komplexes.
Repressoren können diese Interaktion durch ihre eigene Bindung
unterdrücken.
Regulation der Transkription bei Eukaryonten
Die Transkription eines Gens kann auf folgenden Ebenen reguliert
werden:
- Aktivierung des Gens
- Initiation der Transkription
- Prozessing des Transkriptes
- Transport ins Cytoplasma
- Translation der mRNA
Die meisten regulatorischen Proteine setzen an der Transkription an.
Wenn eine Gruppe von Genen gemeinsam kontrolliert wird, findet man bei
allen Mitgliedern ein Response-Elemten, eine identische
Promotorstruktur, die mit einem bestimmten TF interagiert.
Ein beispiel für die Kontrolle einer Gengruppe durch einen Faktor
stellt die Hitzeschockantwort der Zelle da. Bei den Eukaryonten
befindet sich der dafür zuständige Bindebereich ausserhalb der
Promotorregion und wirkt als ,,alternativer`` Promotor für
Proteine, die teilweise auf unterschiedliche ARten durch verschiedene
Proteine kontrolliert werden.
Man findet bei den Eukaryonten häufig Mechanismen, bei denen
unterschiedliche Sequenzelemente voneinander unabhängig dazu in der
Lage sind, das zugehöroge Gen zu aktivieren.
Die Interaktion der DNA geschieht über unterschiedliche DNA-bindende
Motive:
- Steroidrezeptoren
- Zinkfinger-Motiv
- Helix-Turn-Helix-Motiv
- Helix-Loop-Helix-Motiv
- Leucin-Zipper-Motiv
Die DNA-bindenden Motive sind vor allem in den induzierbaren TF zu
finden, die nur in bestimmten Zellen synthetisiert werden und in ihrer
Aktivität moduliert werden können.
Beim Zinkfinger interagiert eine Gruppe von Aminosäuren mit einerm
Zinkion. Dieses Motiv ist das Hauptmotiv der Steroidrezeptoren.
Durch die Interaktion mit dem Zink bilden die AS bei dem
Cys2-His2-Finger einen ,,
Finger`` aus, der mit seinem C-Terminus eine 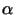 -Helix
ausbildet, die zwei sequenzspezifische Kontakte mit der DNA
eingeht. Diese Struktur bildet sich drei Mal aus. Zusammen passen die
drei Finger in eine grosse Furche. -Helix
ausbildet, die zwei sequenzspezifische Kontakte mit der DNA
eingeht. Diese Struktur bildet sich drei Mal aus. Zusammen passen die
drei Finger in eine grosse Furche.
Bei dem Cys2-Cys2-Finger hingegen findet man häufig nur eienn
Finger.
Bei Glucocoricoid- und Estrogen-Rezeptoren findet man zwei Finger, die
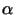 -Helices ausbilden, die über -Helices ausbilden, die über 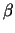 -Faltblätter zu einer
hydrophoben Struktur verbunden sind. Diese Rezeptoren dimerisiern und
bedecken hintereinander zwei Windungen der grossen Furche. -Faltblätter zu einer
hydrophoben Struktur verbunden sind. Diese Rezeptoren dimerisiern und
bedecken hintereinander zwei Windungen der grossen Furche.
Die Steroidhormone wirken in der Zelle über die Bindung an einen
spezifischen Rezeptor. Das Molekül diffundiert in die Zelle und bindet
dort an seinen Rezeptor. Dadurch wird dann der Rezeptor, wie etwa der
Glucocoricoid-Rezeptoren dazu befähigt, eine bestimmte
Consensussequenz zu erkennen und aktiv zu werden.
Die so erkannten Responseelemente folgen einem bestimmten Consensus,
bestehend aus zwei Halbelementen, da der Rezeptor, wie oben erwähnt,
als Dimer bindet. Man bezeichnet diesen Protess als eine
ligandenabhängige Transkription.
Bei Homodimere liegen diese Kopf-an-Kopf und erkennen palindromische
Sequenzen, während bei Heterodimere die Dimerisierung Kopf-an-Schwanz
erfolgt.
Die Homöobox ist eine DNA-Sequenz von 60 AS länge, die in bestimmten
Proteinen der meisten Eukaryonten auftritt. Sie wurde zuerst bei den
für die Entwicklung von Drosophila verantwortlichen Genen entdeckt.
Sie sorgen bei diesen Proteinen für die spezifische Erkennung der
DNA. Sie befindet sich zumeist am C-Terminus und weist eine hohe
Homologie zu den Sequnenzen bei prokaryontischen Repressoren auf.
Eine Dimerisierung kann von speziellen Domänen wie bei den
Steroidrezeptoren oder von dem DNA-bindenden Motiv selbst wie bei dem
HTH-Motiv vermittelt werden. In den meisten HLH-Motiven findet man in
der Nachbarschaft eine basische Region, die für die DNA-Bindung
notwendig ist und deshalb wird das Protein auch als bHLH
bezeichnet. Man teilt die bHLH-Proteine in eine Klasse A die ubiquitär
exprimiert wird und eine Klasse B die gewebespezifisch exprimiert
wird, ein. Letztere sind als Heterodimere besonders aktiv; bei den
ersteren vermutet man eine Homodimerisierung, ist sich aber über die
genaue Struktur nicht klar.
Die Regulation kann bei diesen Proteinen über einer Veränderung der
Dimerisierungs- oder der Bindungseigenschaften geschehen. So können
manche HLH-Proteine auch im Prozess der Dimerisierung als Supressoren
wirken, indem sie funktionsunfähige Dimere bilden. Durch die
unterschiedliche Dimerisierung kann eine geringe Anzahl an Proteinen
eine grosse Variabilität hervorbringen.
Ein ebenfalls dimerisierendes Motiv ist das Leucin-Zipper-Motiv. Es
enthält einen Abschnitt mit Leucinresten, die das Motiv für die
Dimerisierung bereitstellen. Die Leucinreste ragen aus einer
amphipathischen Helix heraus und verzahnen sich miteinander
vergleichbar einen Reissverschluss. Durch die Dimerisierung werden die
DNA-bindenden Region der beiden Untereinheiten zueinander gebracht und
können so mit der DNA interagieren.
Durch den Reissverschluss entsteht eine Y-förmige Konformation,
bZIP-Motiv genannt. Die beiden Arme des Y sind die basischen
DNA-bindenden Sequenzen, die so mit der DNA interagieren können.
In vivo stellt sich die Frage, wie die Transkriptionsfaktoren Zugang
zu der dicht gepackten DNA erhalten. Ein Gleichgewichtsmodell bei dem
die TF mit den Histonen um Bindungsstellen konkurrieren würde die
stabile Transkription in der S-Phase, in der eine grosse Menge an
Histonen existiert, nicht erklären.
Das Okkupationsmodell besagt, dass die Proteinklasse, die sich zuerst
an die DNA anlagert von der anderen nicht mehr verdrängt werden
kann. In vitro-Experimente scheinen dieses Modell zu bestätigen. Dies
bedeutet, dass der Faktor schon bei der Replikation präsent sein muss,
damit die Bindung der Nukleosomen verhindert wird. Ansonsten ist die
DNA dauerhaft blockiert.
In neuerer Zeit hat man herausgefunden, dass die Histone von der DNA
durchaus unter ATP-Verbrauch verdrängt werden können. Dieses
dynamische Modell sieht vor, dass ein TF sich an die DNA anlagert,
unter ATP-verbrauch die Nukleosomen verschiebt und so eine
Nuklease-hypersensitive Region schafft.
Bei einigen TF hat man auch nachgewiesen, dass sie sich an die DNA an
der Oberfläche der Nukleosomen anlagern können. Es gibt sogar
Strukturen, bei denen die Wechselwirkung zwischen dem TF und dem
Chromatin notwendig ist, damit die Transkription startet.
Da die Transkription von menschlichen Genen in der Maus deulich
unterhalb des natürlichen Niveaus liegt, kam man zu dem Schluss, dass
die Regulation neben Enhancer und Promotor auf weiteren Funktionen
aufbauen muss.
Diese Stellen sind DNase-I-hypersensitiv und befinden sich am Ende des
Clusters. An der 5'-Seite findet man beim 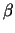 -Globin z.B. eine
hypersensitive locus control region LCR; ob der hypersensitiven
3'-Region eine Funktion zukommt ist ungewiss.
Eine ähnliche Region findet man bei den -Globin z.B. eine
hypersensitive locus control region LCR; ob der hypersensitiven
3'-Region eine Funktion zukommt ist ungewiss.
Eine ähnliche Region findet man bei den 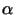 -Globinen und man
vermutet, dass diese Region dazu dient, die DNA zu öffnen. Bei anderen
Genen konnte man diese Sequenz nicht finden. -Globinen und man
vermutet, dass diese Region dazu dient, die DNA zu öffnen. Bei anderen
Genen konnte man diese Sequenz nicht finden.
In vielen Bereichen findet man Isolatoren, die dazu dienen, Gene von
den Einflüssen der Nachbarn zu isolieren. Platziert man einen Isolator
zwischen einem Enhancer und einem Promotor, kann zwischen diesen keine
Wechselwirkung mehr stattfinden.
Bei der Analyse von hsp70 in Drosophila fand man eine Sequnenz scs,
die jegliche regulatorische Wirkung von einer Region auf die nächste
verhindert. Diese findet man vor allem in den Interbanden, was dafür
spricht, dass die Querbanden der Chromosomen funktionelle Einheiten
darstellen, die durch die Interbanden getrennt sind. Diese Wirkung ist
von der Orientierung unabhängig; er unterdrückt allerding immer den
Enhancer, der in der von ihm aus abgewandten Seite des Promotors
liegt. Die Funktionsweise dieses Elements ist noch nicht geklärt.
Zwischen LCR und Isolator kann eine matric attachment site MAR liegen,
die die Domäne an der Kernperipherie verankert.
Bei den Eukaryonten spielt die Methylierung der DNA eine wichtige
Rolle bei der Transkription. In einigen Geweben wird die Aktivität der
Transkription durch eine Methylierung der Gene verhindert.
Ein Gen, das aktiv ist, weil es nicht methyliert ist, bezeichnet man
als untermethyliert.
Man stellt sich vor, dass die TF entweder erst nach dessen
Demethylierung an den Promotor binden oder aber sich durch die
Demethylierung eine Strukturveränderung ergibt, die die Initiierung
der Transkription erst ermöglicht.
Bei manchen Genen findet eine Methylierung auch dann statt, wenn die
Gene stark methyliert sind.
In manchen 5'-Regionen findet man CpG-reiche Inseln, die mit der
Wirkung der Methylierung direkt verknüpft sind. Diese Inseln sind
normalerweise unmethyliert und finden sich bei allen Haushaltsgenen,
sowie bei den Promotoren von gewebeabhängig exprimierten
Genen. Ausserhalb dieser Regionen findet man sie selten. Die
CpG-reichen Inseln deuten demnach darauf hin, dass das Gen potentiell
aktiv ist; reichen allerdings als Transkriptionssignal nicht aus.
Die Methylierung der CpG-Region unterdrückt normalerweise die
Transkription des entsprechenden Gens. Dies beruht sehr
wahrscheinlich auf der Bindung eines Proteins, MeCP-1 an die
methylierten Basen.
Die methylierten Stellen kann man in konstante und variable Regionen
einteilen. Die zuerst genannten werden in allen Geweben methyliert;
letztere liegen in den Geweben, in denen eine Expression stattfindet
demethyliert vor. Im Sperma sind sowohl die konstanten als auch die
variablen Bereiche methyliert und man vermutet, dass das Fehlen einer
Methylierung auf deren Verlust zurückzuführen ist.
Ob es eine Neumethylierung von nicht exprimierten Genen gibt ist
ungewiss.
Man nimmt an, dass die Methylierung weiter gegeben wird, indem die
Methylase an dem hemimethylierten Material aktiv wird und den nach der
Replikation nicht methylierten Strang methyliert.
Auf diese Weise kann auch in der Keimzelle ein spezifisches Muster von
Methylgruppen angebracht sein, das als genetisches imprinting weiter
gegeben wird.
So hängt bei Mäuseembryos die Expression einiger Gene von dem
Geschlecht des Elternteils von dem sie stammen ab. Dieses Muster wird
während der Gametogenese geprägt und wird das gesamte Leben über
beibehalten. Einzig in den Urkeimzellen des neuen Tieres findet eine
Veränderung des Musters de novo statt.
|





