Unterabschnitte
Das Replicon
Einer Zellteilung muss immer eine Verdoppelung der DNA, die
Replikation, vorausgehen. Die DNA-Einheit, an der ein
Replikationsvorgang abläuft wird als Replicon bezeichnet. Das Replicon
besitzt einen Replikationsursprung oder origin und einen Terminus, der
die die Replikation beendet.
Jedes DNA-Molekül, das ein origin enthält kann autonom repliziert
werden. Wenn die Replikation erst einmal begonnen hat, wird sie erst
beendet, wenn das gesamte Genom verdoppelt ist. Während dieses
Prozesses darf keines der Replikons mehrfach repliziert werden.
Die Replikation beginnt immer mit der Bildung einer Replikationsgabel
am origin. Wenn sich eine Replikationsgabel bildet, verläuft die
Replikation uni- ansonsten bidirektional. Die Replikation öffnet die
DNA und macht unter dem EM ein Auge, eine sogenannte
Theta-(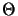 )-Struktur sichtbar. Ob die Replikation uni- oder
bidirektional verläuft lässt sich durch zwei radioaktive Markierungen
nacheinander nachweisen. Wenn beide an einem Ende des Auges auftreten
ist die Replikation uni- ansonsten bidirektional. )-Struktur sichtbar. Ob die Replikation uni- oder
bidirektional verläuft lässt sich durch zwei radioaktive Markierungen
nacheinander nachweisen. Wenn beide an einem Ende des Auges auftreten
ist die Replikation uni- ansonsten bidirektional.
Das bakterielle Genom ist ein einziges ringförmiges Replicon. Den
Replikationsursprung der Bakterien konnte man isolieren, indem man die
genomische DNA wahllos in Plasmide ohne einen origin einbrachte. Der
Plasmid, in dem sich der Ursprung befand, konnte sich als einzige
replizieren.
Das Genom von E. coli wird bidirektional von einem Ursprung, dem oriC
aus repliziert. Durch die Ringform umgehen die Prokaryonten das
Problem, die Enden der DNA zu replizieren.
Die oriC-sequenz ist zwar für die Initiation der Replikation, nicht aber
für die Seggregation der DNA verantwortlich. Diese wird durch Enzyme
vermittelt.
Die beiden Terminationssequen von E. coli (wegen der
Bidirektionalität) befinden sich ca. 100 kbp voneinander entfernt,
wobei sich die so entstehenden Bereiche überlappen. Dies hat zur
Folge, dass die beiden sich normalerweise auf halber Stecke treffenden
Polymerasen in dieser Region auch dann wenn die ander noch nicht
angekommen ist, anhalten.
Wenn die DNA-Polymerase bei der Replikation auf eine RNA-Polymerase
trifft, die in gleicher Richtung die DNA abliest, kann diese durch
einen noch ungeklärten Mechanismus ,,überholt`` werden. Bewegt
sie sich in anderer Richtung (was allerdings nur selten vorkommt),
dann kann es zu einem Stop der Replikation kommen. Der genaue
Mechanismus ist noch unbekannt.
In den Zellen der Eukaryonten findet eine Replikation nur in der
S-Phase statt. Dabei gibt es auf jedem Chromosom viele
Replicons. Diese sind kleiner als die der Eukaryonten und schreiten
langsamer voran. Ausserdem ist der Ablauf ihrer Aktivierung
kontrolliert, so dass bestimmte Bereiche des Chromosoms ,,früh
replizierend`` und andere ,,spät replizieren`` sind. Sehr
wahrscheinlich sind zu jedem Zeitpunkt nur etwa 15% der Replicons
aktiv. Man nimmt an, dass dabei keine Terminatoren existieren, sondern
die Replikation durch das Aufeinandertreffen zweier Replikationsgabels
zu Stande kommt. Wie das Chromosom verhindet, das bereits replizierte
DNA repliziert wird, ist unklar.
Bei Hefe konnte man mit einer Anlyse von Plasmiden eine ARS (Autonom
replizierende Sequenz) eine Sequenz identifizieren, die für eine
effiziente autonome Replikation von Plasmiden notwendig sind. Diese
ARS-Sequenz entspricht dem Replikationsursprung und ist sehr
wahrscheinlich durch eine Consensussequenz, vor allem aber durch
AT-reiche Banden gekennzeichnet.
Man vermutet, dass dem Replikationsstart in den höheren Eukaryonten
noch wesentlich kompliziertere Mechanismen zu Grunde liegen.
Während man bei den Eukaryonten keine Replikatinsursprünge finden
konnte, hat man bei deren Mitochondrien einen Replikationsursprung
entdeckt. Dort schreitet die Replikation zunächst ein kleines Stück
weit an einem Strang entlang und verdrängt dort den Partner. So
entsteht eine D- oder Verdrängungsschleife.
Bei den Mitochondrien der Säuger wird dieser Bereich dann weiter
ausgedehnt und verdrängt den anderer (L-)Strang immer mehr. Nachdem
zwei Drittel des L-Strangs frei gelegt wurden, wurde dann auch dessen
Ursprung freigelegt, an dem dann die Replikation eines neuen H-Strags
beginnt. Das Modell wird als rolling circles bezeichnet.
Ein Problem bei der Replikation linearer DNA sind deren Enden. Da die
Polymerase sich immer nur in einer Richtung bewegen kann müsste sie an
einem Ende der DNA ganz aussen an derem Ende binden. Es ist unklar, ob
dies möglich wäre.
Bei einigen Viren wie dem Adenovirus wird dieses Problem durch
Strangverdrängung gelöst. Dabei verdrängt die Synthese des einen
Strangs den anderen. Dieser bildet in der einzelstängigen Form mit
seinen Enden einen doppelsträngigen Ursprung und wird auf diese Art
und Weise repliziert. Bei diesen Mechanismus wird ein sogenanntes
terminales Protein verwendet. Dieses bindet an das Ende der DNA, das
ein Nukleotid als Primer gebunden hat und die Bindung der Polymerase
vermittelt.
Bei einem anderen Mechanismus, dem rollenden Ring wird nur einer der
beiden Stränge repliziert. Nachdem ein Einzelstrangbruch den Ring
öffnet, wird das dadurch entstehendes freie 3'-Ende von der Polymerase
verlängert. Mit dieser Verlängerung fährt sie fort, so dass ein
multimerer Einzelstrang entsteht. Dieser Mechanismus findet z.B. bei
der Amplifikation der rRNA von Xenopus eine Rolle, vor allem aber
findet man ihn bei Bakteriophagen.
Bei Bakterien wird bei der Konjugation ein Plasmid auf ein anderes
Bakterium übertragen. Diese Konjugation wird von einem sogenannten
F-Plasmid vermittelt. Dieses Episom hat die Möglichkeit, sich entweder
autonom zu replizieren oder mit dem Genom zusammen repliziert zu
werden. Ein F-positives (mit F-Plasmid) Bakterium kann mit einem
F-negativen konjugieren. Bei der Konjugation wird der Plasmid und in
einigen Fällen sogar das gesamte Genom übertragen.
Eine bestimmte Region des Plasmids, die Transferregion steuert den
DNA-Transfer. Bei F-positiven Bakterien finden sich an der Oberfläche
sogenannte Pili, die vom F-Faktor gesteuert werden und deren Kontakt
mit einer anderen Zelle die Paarung auslöst.
Der F-Plasmid wird dann in zwei Einzelstränge aufgetrennt und einer
dieser Stränge wird an die andere Zelle weiter gegeben. In beiden
Zellen werden die Einzelstränge dann wieder zu einem Doppelstrang
ergänzt.
Bei einem in das Genom integrierten F-Plasmid werden neben dem
eigentlichen Plasmid auch weite Teile des Genoms übertragen. Dieser
integriert sich in das Genom der Akzeptorzelle. So können
die Bakterien genetische Informationen austauschen.
Bei Bakterien erfolgt die Häufigkeit der Replikation in Korelation zum
Wachstum der Zelle und nach einer abgeschlossenen Replikation kommt es
immer zu einer Zellteilung. Die Geschwindigkeit der Zellteilung ist
von der Geschwindigkeit der Replikation und der Zeit, in der sich die
Komponenten, die für die Teilung erforderlich sind, zusammenlagern.
Da eine zweite Replikation schon vor dem Ende der ersten eingeleitet
wird, entstehen bei der Replikation der Bakterien mehrfach gegabelte
Chromosomen. Das Startsignal für die Replikation ist das Erreichen
eines bestimmten Verhältnisses von Chromosomenursprüngen zur
Zellmasse. Sehr warscheinlich ist das eigentliche Signal entweder die
Ansammlung eines Initiators oder die Verdünnung eines Inhibitors.
Die eigentliche Teilung erfolgt durch die Ausbildung eines Septums,
einer Einstülpung der Membran in der Mitte der Zelle. Bevor sich das
Septum ausbildet, bildet sich der Annulus um die Zelle herum. An
dieser Struktur wird der Kontakt zwischen Membran und Zellwand
gefördert. Diese Struktur findet man auch bei einer neu gebildeten
Zelle. Bei der Teilung wird das Annulus durch die Bildung des Septums
ersetzt und in der Nachbarschaft bilden sich zwei neue Annuli.
Man vermutet, dass das Gen ftsZ die zellteilung initiiert. Mutanten in
diesem Gen haben einen Defekt in der Bildung des Septums. Ein Septum
kann sich in der Zelle an drei Punkten ausbilden: in der Mitte der
Zell und an den Polen. An den Polen wird dies verhindert, indem ein
Gen minE einem anderen Genprodukt (minCD), das als Inhibitor wirkt, in der
Mitte entgegenwirkt und so nur dort eine Ausbildung des Septums
gestattet. Das Verhältnis beider Proteine entscheided also über die
Bildung des Septums.
Die Verteilung der Chromosomen erfolgt durch den aktiven Transport
durch einen Proteinkomplex. Man findet dazu in der Zelle ein
Anheftungsprotein an die Membran, MukA und ein dem Dynein verwandtes
Protein MukB.
Bei der Plasmidreplikation unterscheidet man ein
Einzelkopie-Kontrollsystem, bei dem die Replikation der gleichen
Kontrolle wie auch die Chromosomen unterliegt und ein
Mehrfachkopie-Kontrollsystem, bei dem sich ein Plasmid in einer Zelle
auch mehrfach replizieren kann.
Bei der Replikation muss durch ein ortsspezifisches
Rekombinationssystem sichergestellt werden, dass keine Dimerisierung
der neu synthetisierten Ringe statt findet oder diese wieder gelöst
wird.
Durch ein Anhängigkeitssystem wird gewährleistet, dass das Bakterium
nur zusammen mit dem Plasmid überleben kann. Dies kann zum Beispiel
durch ein stabiles Gift und ein labiles Gegengift erreicht
werden. Verliert das Bakterium den Plasmid, wird das Gegengift
schneller abgebaut und das Bakterium stirbt.
Bei Plasmiden mit einer geringen Kopienzahl gibt es einen Mechanismus,
der reguliert, dass sich bei einer Zellteilung die Plasmide auf die
beiden Seiten des Septums aufteilen. Dies könnte auf einer begrenzten
(eventuell nur zwei) Anahl von Bindungsstellen oder einen der
Chromosomenverteilung ähnlichen Mechanismus, bei der sich die Plasmide
in Paaren anordnen, zurückzuführen sein.
Mitglieder einer Kompatibilitätsgruppe von Plasmiden können in einer
Zelle nicht zusammen existieren, da sie z.B. bei der Trennung nicht
unterschieden werden können. Man nimmt an, dass die Regulation der
Kopienanzahl durch einen Repressor erfolgt, der die Anzahl der
Replikation-origins misst. Dieser Mechanismus würde dann bei zwei
unterschiedlichen Plasmiden mit dem selben origin ebenfalls greifen,
die Replikation verhindern und dazu führen, dass die Plasmide ungleich
über die Zellen verteilt werden.
Die Regulation der Kopienanzahl pro Zelle kann z.B. wie im Fall des
Co1E1-Plasmid über die Kontrolle eines RNA-Primers geschehen. Zum
einen wird die Verfügbarkeit dieses Primers über die Verbindung mit
einem Gegentranskript gehemmt, zum anderen durch ein Protein, das in
der Nähe des Primers kodiert ist, beeinflusst.
|





