Unterabschnitte
Die Replikation
Für die Replikation der DNA bei E. coli sind insgesamt drei
unterschiedliche Polymerasen, mit Polymerase I bis III bezeichnet,
verantwortlich. Alle bakteriellen Enzyme weisen
eine 3'-5'-Exonuklease, die entgegen der Reichtung, in der die
Polymerase arbeitet ein Korrekturlesen (proof-reading) vornimmt und
falsche Basen entfernt, auf. Bei einer falschen Basenpaarung bewegt
sich die Polymerase wieder ein Stück zurück und löst die falsch
gepaarte Base ab.
Die Polymerase I hat die Fähigkeit, die Replikatin in vitro an einem
Einzelstrangbruch zu beginnen. Während an dem Bruch (nick) neue DNA
synthetisiert wird, kann dadurch die alteDNA verdrängt werden. Dieser
Prozess wird Nicktranslation genannt. Sie dient in vivo wohl vor allem
dem Auffüllen von kurzen einzelsträngigen Bereichen.
Die eigentliche Replikation wird von der Polymerase III
durchgeführt. Die Fehler, die die Polymerase machen kann, können
entweder in dem Einbau des falschen Nukleotids liegen oder es kommt
durch den Einbau von einem zusätzlichen, bzw. dem Auslassen von einem
Nukleotid zu einer Verschiebung des Leserasters. Die beiden
Fehlertypen werden unterschiedlich korrigiert:
Die Wahrscheinlichkeit einer Leserrasterverschiebung wird durch die
Prozessivität der Polymerase beeinflusst. Unter Prozessivität versteht
man die Tendenz des Proteins auf einer einzelnen Matrize zu
verbleiben, statt sich zu lösen. Je höher die Prozessivität desto
geringer das Auftreten von Leserasterverschiebungen.
Die Korrektur von falschen Basen erfolgt durch die
Exonuklease-Aktivität.
Bei Eukaryonten findet man zwei DNA-Polymerasen, die Polymerase
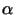 und die Polymerase und die Polymerase 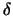 .
Die DNA-Polymerase .
Die DNA-Polymerase 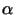 initiiert die Bildung des Strangs, während initiiert die Bildung des Strangs, während
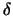 den Strang verlängert. (Folgestrang, Leistrang ???) den Strang verlängert. (Folgestrang, Leistrang ???)
Die DNA-Polymerase ist immer auf einen Primer zur Initiierung der
Replikation angewiesen. Dieser kann aus einerm Stück RNA bestehen, er
kann ein Stück DNA (z.B. nach einem Einzelstrangbruch) sein oder von
einem Protein, das ein Nukleotid gebunden hat, gebildet werden.
Man unterscheidet bei der Replikation einen Leitstrang, der
kontinuierlich von 5' nach 3' synthetisiert wird und einen
Folgestrang, der Stückweise ebenfalls von 5' nach 3' gebildet wird.
Im Folgestrang werden Okazaki-Fragmente mit einer Länge von 1000 bis
2000 bp synthetisiert, die dann kovalent miteinander Verbunden
werden. Sowohl bei Eukaryonten, wie auch bei E. coli findet man die
semidiskontinuierliche Replikation. Die Primer zum Beginn der
Polymeraseaktivität bzw. der -aktivitäten auf dem Folgestrang besteht
aus RNA. Der Primer wird nach der Replikation entfernt und durch die
Polymerase I aufgefüllt und durch die DNA-Ligase verbunden.
Die Bildung der Replikationsgabel bedarf zunächst zweiter Proteine,
der Helikase, die die DNA-Stränge trennt und der einzelstrangbindenden
Proteine, die eine Zusammenlagerung der gertrennten Einzelstränge
verhindern. Die Bindung dieser Moleküle erfolgt kooperativ und bedeckt
so die gesamte einzelstängige DNA.
Bei E. coli initiiert der Aufbau eines Primosoms an der
Primosomenaufbaustelle die Replikation. Das Primosom bewegt sich von
einer Bindungsstelle für die Primer der Okazaki-Fragmente zur
nächsten.
Das Anbringen der Primer ist Aufgabe der Primase. Dabei handelt es
sich um eine RNA-Polymerase, die ein kurzes RNA-Stück als Primer
synthetisiert.
Bei E. coli hat man Terminationssequenzen von 23 bp länge gefunden,
die die Replikation in einer Richtung aufhalten, allerdings sehr
wahrscheinlich durch eine aus der anderen Richtung kommende Polymerase
verdrängt werden können.
Die Koordination der Replikation von Leit- und Folgestrang wird auf
unterschiedliche Arten gewährleistet:
Man nimmt an, dass die beiden Polymerasen der Prokaryonten für den Leit- und
Folgestrang ein Holoenzym bilden, das der Helikase folgt. Während die
Polymerase des Leistrangs diesem kontinuierlich folgt, bildet der
Folgestrang durch die Aktivität der anderen Polymerase eine Schleife
aus, in der bereits der Primer aufgebracht wird, so dass die andere
Polymerase, sobald sie sich von dem Folgestrang löst, sofort mit der
Synthese des nächsten Framentes beginnen kann.
Bei den Eukaryonten liegen zwei Polymerasen, die Polymerase
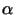 und die Polymerase und die Polymerase 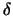 vor. Früher dachte man, dass die
Polymerase vor. Früher dachte man, dass die
Polymerase 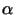 den Folgrstrang und die Polymerase den Folgrstrang und die Polymerase 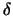 den
Leitstrang synthetisiert; nun scheint es aber so, dass die Polymerase den
Leitstrang synthetisiert; nun scheint es aber so, dass die Polymerase
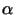 für die Inititation der Replikation verantwortlich ist, indem
sie den RNA-Primer um ein kurzes Stück DNA verlängert, an dem dann die
Polymerase für die Inititation der Replikation verantwortlich ist, indem
sie den RNA-Primer um ein kurzes Stück DNA verlängert, an dem dann die
Polymerase 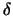 angreifen kann. Es scheint als wäre die Polymerase angreifen kann. Es scheint als wäre die Polymerase
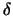 für die Elongation beider Stränge verantwortlich. für die Elongation beider Stränge verantwortlich.
Die DNA des Phagen unterscheidet sich von der DNA der Zelle dadurch,
dass der Phage Hydorxymethylcytosin an der Stelle von Cytosin
verwendet und so seine DNA von der des Wirts unterscheiden kann.
Die Replikation des Phagen wird von einem Multienzymkomplex
katalysiert.
Die Replikationsmaschinerie gleicht vom Aufbau der des Wirtes, nur
wird nicht dessen Maschinerie benutzt, da die so hergestellte DNA zu
schnell abgebaut würde.
Es scheint so, dass allen Systemen die Probleme der REplikation gemein
sind und sie auf ähnliche Art und Weise gelöst werden:
- Die Helikase öffnet die DNA
- Die Primase bringt einen Primer an
- Einzelstranbindende Proteine stabilisieren die Einzelstränge
- Eine gleitende Klammer hält den Komplex an der DNA
- Diese Klammer wird durch eine ATPase beladen
- Die eigentliche Katalyse muss durchgeführt werden
- In vielen Fällen dimerisieren die Untereinheiten der Polymerase
- Die Primer-RNA muss entfernt werden
- Die Ligation wird von einer Ligase geschlossen.
- In vielen Fällen verhindert eine Topoisomerase eine
Überspiralisierung
Bei der Entstehung der Replikationsgabel bei E. coli sind sechs
Proteine beteiligt: DnaA, DnaB, DnaC, HU, Gyrase und SSB.
DnaA ist nur an der Initiation beteiligt und bindet als erstes Protein
an die oriC-Sequenz. Dann schmilzt es die DNA an mehreren auf den oriC
folgenden Stellen auf und sorgt dafür, dass diese einen offenen
Komplex bilden.
DnaB und DnaC bilden den Motor der Initiation, vergleichbar dem
Primosom. Ausserdem weist der Komplex aus diesen Proteinen
Helikase-Ativität auf. Dieser Komplex bindet an die durch DnaA
erzeugte Einzelstrangstruktur. Die Helikasereaktion ist ATP-abhängig.
Die Gyrase dient als Drehgelenk und verhindert eine
Überspiralisierung. Die SSB stabilisieren die Einzelstränge und das
Protein HU ist eine DNA-bindendes Protein, das die DNA verbiegen kann
und sehr wahrscheinlich der Strukturbildung dient.
Nachdem die sich die Replikationsgabel gebildet hat, findet die erste
Primingreaktion statt. Das Priming wird durch das Aufschmelzen der DNA
eingeleitet.
In Bakterien hängt die Initation der Replikation von der Methylierung
des oriC ab. Im Normalzustand ist die DNA methyliert. Wenn nun die
Replikation statt gefunden hat liegt die DNA in einer hemimethylierten
Form vor, in der sie nicht weiter repliziert wird. Die Sequenz wird
erst nach ungewöhnlich langen Zeit von 13 Minuten wieder methyliert.
Diese Verzögerung wird von einem speziellen Protein SeqA induziert,
indem es stark an die hemimetyhlierte DNA bindet und diese so für die
Methylierung blockiert.
Eine weitere Eigenschaft der hemimethylierten DNA ist eine gute
Bindung an die Zellmembran. Dies könnte eine Reinitiation der
Replikation verhindern. Die Anheftung an die Membran könnte eine
Funktion von DnaA sein.
In einer Eukaryontenzelle muss gewährleistet sein, dass die
Replikation an jedem Replicon nur einmal stattfindet. Man vermutet,
dass sich im Zellkern ein sogenannter Lizenzfaktor befindet, der
während der Replikation aufgebraucht wird und nur dadurch, dass
in der Mitose neuer Faktor aus dem Cytoplasma in den Zellkern gelangt
wieder reaktiviert werden kann. Ein potentieller Kandidat für diesen
Mechanismus wären die MCM2, 3 und 5-Proteine, die man in der Hefe
nachweisen konnte.
|





