Unterabschnitte
Zellkern
Bei den Eukaryonten ist die DNA im Zellkern eingschlossen. Der Kern
nimmt rund 10% des Zellvolumens ein und wird gegen das übrige
Cytoplasma durch die aus zwei Membranen bestehende Kernhülle
abgegrenzt. Sie geht dann direkt in das Endoplasmatische Retikulum
über.
Die Kernporen dienen dem aktiven Transport von Proteinen in das
Cytoplasma.
Die Kernhülle wird von zwei Netzwerken aus Intermediärfilamenten
gestützt. Eine davon stützt als Kernlamina die Membran von Innen, die
andere weniger geordnet von aussen.
Es ist noch strittig, wie im Zellkern eine Kernmatrix aufgebaut
ist. Nach einer Reihe von Extraktionsschritten bleiben bestimmte
DNA-bindende Proteine, die SARs oder MARs übrig. Man postuliert, dass
diese Strukturen die Schleifen der Chromosomen bilden und für deren
Ausrichtung im Inneren des Kerns verantwortlich sind.
Die Aufgabe des Zellkerns könnte darin bestehen, entweder die DNA vor
den mechanischen Kräften des Cytoskeletts zu schützen oder aber das
Splicen der RNA zu ermöglichen ohne dass es dabei zu Konflikten mit
einer eventuell bereits beginnenden Translation kommt.
Chromosomen
Jedes DNA-Molekül wird in einem Chromosom verpackt.
Um sich zu replizieren und so an die Tochterzellen weiter zu geben,
benötigt ein Chromosom einen Replikatinons-Startpunkt (siehe
3.1). Des weiteren ist eine bestimmte Sequenz, das
Centromer notwendig, um die DNA bei der Zellteilung an der
Mitosespindel zu befestigen.
Das Telomer bildet die dritte
charakteristische Sequenz eines jeden Chromosoms. Sie befindet sich am
Ende und dienen dazu, das Problem, dass man für die Endstücke der DNA
keine Matrix findet und diese deshalb bei der Replikation verkürzt
werden, zu umgehen. dazu hängt die Telomerase periodisch Nukleotide an
die Sequenz an kompensiert so den Verlust von DNA.
Der Telomer besteht aus einer Wiederholung einer kurzen Sequenz (beim
Menschen GGGTTA), die bei allen Eukaryonten sehr G-reich ist. Die
Telomerase enhält einen kurzen RNA-Strang, der als Matritze für die
Bildung neuer DNA dient.
Bei den höheren Eukaryonten ist ein Gen aus Introns und Exons
aufgebaut. Das primäre RNA-Transkript, das direkt von den Gen
abgelesen wird, wird durch Splicen, d.h. Entfernen der Intronsequenzen
in die mRNA überführt.
Die DNA-bindenden Proteine werden in Histone und Nicht-Histone
eingeteilt und bilden zusammen mit der DNA das Chromatin.
Die Histone binden durch ihre strake positive Ladung an die DNA. Sie
sind kleine Proteine und lassen sich in Nikleosomen-Histone, die die
DNA in Nukleosomen aufwickeln und die H1-Histone, die grösser und
weniger konserviert sind.
Die Nukleosomen bilden die Grundeinheit des Chromosoms. Diese bestehen
aus acht Nukleosomen-Histonen, um die die DNA dikusförmig aufgewickelt
ist. Der Histon-Kern wird als Histon-Oktamer bezeichnet.
Die H1-Histone binden an den Nukleosomen und nehmen gleichzeitig zu
dem folgenden Nukleosom Kontakt auf, so dass diese zu einer geordneten
Struktur finden.
Die Stellen, an denen die DNA weniger dicht gepackt ist, werden von
sequenzspezifischen DNA-bindenden Molekülen bedeckt.
In der Meiose bilden die Chromosomen eine Struktur aus, in der sie
Lampenbürstenchromosomen genannt werden. Sie bilden dabei Schleifen
aus, an denen eine sehr intensive RNA-Synthese stattfindet. Das nicht
in Schleifen vorliegende Chromatin, die Chromomeren werden nicht
transkribiert.
Diese Chromosomen verdeutlichen, dass das Chromatin nur in gestreckter
Form aktiv, bei dichter Packung hingegen inaktiv ist.
Das aktive Chromatin, an dem RNA synthetisiert wird oder bald
synthetisiert werden soll, kann man durch Verdau mit DNase I
nachweisen.
Ein anderer Fall, in dem die Struktur der Chromosomen gut sichtbar
ist, ist bei den Polytän-Chromosoemen bestimmter Fliegenzelllen. Diese
Zellen enthalten ein vielfaches der normalen DNA, alerdings mit Seite
an Seite liegenden Chromosomen (also nicht polyploid), die dann als
Polytän-Chromosomen bezeichnet werden. Bei diesen Chromosomen kann man
Banden und Zwischenbanden ausmachen, die aus jeweils der gleichen
Sequenz - nebeneinander angeordnet - bestehen, wobei die
Packungsdichte der Banden stärker kondensiert ist.
Durch Entfaltung der Chromatinbande entstehen sogenannte Puffs, an
denen die zu dem Zeitpunkt benötigten RNAs transkribiert werden.
Man nimmt an, dass auch bei diesen Chromosomen das Chromatin in
Schleifenform angeordnet ist und sich in den Puffs dekondensiert.
In den Interphasekernen der höheren Eukaryonten kann man das strak
kondensierte Heterochromatin von dem weniger strark kondensierten
Euchromatin unterscheiden. Das Heterochromation ist
transkriptionsinaktiv. Diese Strukturen sind normalerweise zu fein und
zu gestreckt, als dass man die sehen könnte.
In der Mitose hingegen werden die Chromosomen (wahrscheinlich durch
phosphorylierung des Histons H1) dichter gepackt, damit die ohne zu
brechen auf die Tochterzellen aufgeteilt werden können.
Die Darstellung der 46 Chromosomen wird als Karyotyp bezeichnet. So
kann man die Chromosomen durch Farbstoffe, die gezielt an A-T- oder
G-C-reichen Banden binden, sichtbar machen und ein charakteristisches
Muster darstellen.
Dadurch, dass man ein beliebiges Fragment der Hefe-DNA mit einem
Resistenzgen verbindet und in eine Hefe einbringt, hat man sogenannte
autonom replizierende Sequenzen entdeckt, die den Startpunkt der
Replikation bilden.
Ein in vitro System der Replikation ist bei dem Chromosom des
Affenvirus SV40 gelungen. Bei diesen System wird die Replikation von
einem T-Antigen des Virus eingeleitet. Dieses öffnet eine sogenannte
Replikationsblase, in der der Replikationsapparat der Wirtszelle
angreifen kann.
Bei diesen Vesuchen aht man herausgefunden, dass bei höheren
Eukaryonten zwei Polymerasen existieren: die Polymerase 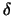 für
den Leit- und die Polymerase für
den Leit- und die Polymerase 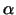 für den Folgestrang. für den Folgestrang.
Der genaue Ablauf der Replikation ist indes noch nicht bekannt.
Bei den Eukaryonten findet man mehrere Startpunkte der Replikation, an
denen diese parallel stratet. Man findet sie gehäuft in sogenannten
Replikationseinheiten. Die verschiedenen Abschnitte des Chromosoms
werden in einer reproduzierbaren Reihenfolge nacheinander repliziert,
wobei das aktive Chromatin früher als das stark kondensierte
repliziert. Die sich spät replizierenden Bereiche entsprechen den
A-T-reichen Banden, die sich - wie oben erwäht - anfärben lassen.
Bei der Replikation müssen enweder Aktivatoren von der DNA entfernt
werden, oder Inhibitoren angebracht werden, damit eine doppelte
Replikation an einem Startpunkt verhindert wird.
Während der S-Phase werden von mehreren Kopien im Genom neue Histone
synthetisiert. Diese werden eventuell von einem speziellen ,,
DNA-Verpackungskomplex``, der hinter der Replikationsgabel
wandert, an die neu gebildete DNA angeheftet.
RNA-Synthese
Die Transkription einer RNA beginnt, wenn die RNA-Polymerase an eine
Promotorregion bindet. Dann werden die beiden DNA-Stränge getrennt und
die Polymerase verlängert schrittweise den RNA-Strang.
Die RNA-Polymerase von E. coli besteht aus vier verschiedenen
Untereinheiten, wobei die 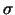 -Untereinheit dazu dient, an den
Promotor zu binden. Diese Untereinheit fällt nach der Synthese der
ersten acht Nukleotide ab und wird durch Elongationsfaktoren
ersetzt. Diese sind wichtig für die Kettenverlängerung. -Untereinheit dazu dient, an den
Promotor zu binden. Diese Untereinheit fällt nach der Synthese der
ersten acht Nukleotide ab und wird durch Elongationsfaktoren
ersetzt. Diese sind wichtig für die Kettenverlängerung.
Einige Bakterien verwenden unterschiedliche 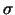 -Untereinheiten,
die zu unterschiedlichen Entwickungszeitpunkten hergestellt werden und
die Polymerase zur Transkription unterschiedlicher Gene veranlasst. -Untereinheiten,
die zu unterschiedlichen Entwickungszeitpunkten hergestellt werden und
die Polymerase zur Transkription unterschiedlicher Gene veranlasst.
Bei den Eukaryonten sind unterschiedliche RNA-Polymerasen für die
Transkription unterschiedlicher Gene verantwortlich. Die
RNA-Polymerasen sind alle sehr komplex aus ca. 10 Untereinheiten
aufgebaut. Die RNA-Polymerase II transkribiert RNA, deren Gene in
Proteine umgesetzt wird, die RNA-Polymerase I transkribiert die
ribosomale RNA und die RNA-Polymerase III macht die kleinen stabilen
RNAs, die die tRNAs und die snRNPs und die kleinen ribosomalen
Untereinheiten.
Wenn man den Zellkern isoliert und auf einem Grid ausbreitet, dann
kann man unter dem Elektronenmikroskop die Chormosomen in einer
perlschnurartigen Weise ausgebreitet betrachten.
Durch diese Untersuchungen fand man heraus, dass die RNA-Polymerase II
bestimmte Gene häufiger trankribiert und sie dabei - wie bei den
Prokaryonten - an einer bestimmten Stelle angreift.
Die so entstandene hnRNA (heterogenous nuclear RNA) wird durch eine
sogenannte Cap, die aus einem methylierten G-Nukleotid besteht,
geschützt. Es schützt vor Abbau und spielt bei der Proteinsynthese
eine wichtige Rolle. Diese Cap wird am 5'-Ende angebracht. Am 3'-Ende
wird das wachsende Transkript geschnitten und mit einem Poly-A-Schwanz
versehen. Der Schwanz dient dem Transport aus dem Zellkern,
stabilisiert die RNA im Cytoplasma und scheint ein Erkennungssignal
für die Ribosomen darzustellen. Diese könnten an 5'-Cap und 3'-Poly-A
erkennen, ob eine RNA intakt ist.
Diese Modifikationen treten bei den andern RNA-Polymerasen nicht auf.
Die hnRNA wird sofort nach ihrem Entstehen in einen den Nuklesomen
ähnlichen hnRNP-Partikel verpackt.
An einigen Stellen findet man ganz besonders stabile Partikel, denen
man eine Rolle beim Splicen der RNA zuordnet und deshalb Splicosomen
nennt.
Die kleinen dafür verantworlichen
Proteinkomplexe werden small nuclear ribonuclear proteins (snRNPs)
genannt. Diese sind mit einigen RNA-Molekülen verbunden und bilden mit
der hnRNA das Splicosom.
Die Sequenz eine Introns ist nebensächlich; die einzigen wichtigen
Bereiche sind die Sequenzen, an denen das Intron entfernt wird. Diese
Stellen bezeichnet man als Donator- (an der 5'-Stelle) und als
Akzeptorstelle (die 3'-Stelle).
Beim Splicen wird die RNA zunächst an der 5'-Stelle von einem U1 snRNP
gebunden und mit an anderer Stelle bindenden Untereinheiten des
Splicosoms zusammengezogen, so dass das Intron eine Schlaufe bildet.
Dann wird die 5'-Stelle geschnitten und an die RNA vor dem 3'-Ende
angehängt, so dass eine Lassostruktur entsteht. In einem leztzen
Schritt wird die 3'-Splicestelle geschnitten, mit der offenen
5'-Stelle ligiert und die Lariat-(Lasso-)Struktur abgespalten.
Wie gewährleistet wird, dass immer die richtigen Stellen verbunden
werden und keine funktionell wichtigen Abschnitte verloren gehen, ist
unbekannt.
Um den Bedarf der Zelle mit Ribosomen zu gewährleisten, enthält das
menschliche Genom ca. 200 Kopien für die rRNA. Die von der
RNA-Polymerase I produzierte 45S-rRNA wird, bevor sie den Zellkern
verlässt, in eine 28S-, enie 18S- und eine 5,8S-rRNA gespalten. So
wird sichergestellt, dass die Untereinheiten im gleichen
Mengenverhältnis hergestellt werden.
Die 5S-rRNa wird getrennt von den anderen Einheiten von der
RNA-Polymerase III transkribiert.
Die rRNA wird im Nukleolus von der RNA-Polymerase I transkribiert und
bereits mit einem proteinreichen Korn am 5'-Ende versehen. Die fertig
transkribierte 45S-rRNA wird in einen Komplex von aus dem Cytoplasma
importierten Proteinen eingehüllt und verliert darin einen Teil seiner
RNA und auch der Proteine und wird dann zu einem
Ribonukleoproteinpartikel zusammengebaut. Nach einer halben Stunde
werden die ersten Untereinheiten mit der 18S-rRNA in das Cytoplama
transportiert, wo dann der Zusammenbau der Ribosomen erfolgt.
Der Nukleus wird nicht von einer Membran umgrenzt, sondern durch die
Kräfte zwischen den ribosomalen untereinheiten
zusammengehalten. Während der Mitose wird der Nukleus aufgelöst und
scheint über die Oberfläche der Chromosomen verteilt auf die
Tochterzellen verteilt zu werden.
Evolution des Kerngenoms
Wenn eine Gensequenz in einer Folge doppelt vorliegt, dann spricht man
von einem Tandem. Dieses kann dann durch weitere Rekombination der
Schesterchromosomen mehrfach hintereinander gehängt werden. Wenn dies
einen Selektionsvorteil schafft (etwa bei den Kopien der rRNAs), wird
diese Veränderung beibehalten.
Durch einen kontinuierlichen Austausch und Genkonversion werden auch
die Bereiche zwischen den codierenden Sequenzen konserviert.
Den Prozess mehrfacher duplikation und schliessender Punktmutation
kann man bei der Enstehung des Hämoglobins und seiner fötalen
Untergruppen gut nachvollziehen.
Bei solchen Rekombinationen und Genduplikationen können auch
Pseudogene entstehen, die nicht aktiv sind und einen grossen Teil des
Säugergenoms ausmachen.
Die Bildung von Hybridproteinen aus vormals getrennten Untereinheiten
und die funktionelle Neukombination von Proteindomänen wird durch die
Introns begünstigt, da so ein wesentlich grösserer Raum für ein
Crossing-Over zur Verfügung steht.
Es ist wahrscheinlich, dass auch bei den Vorfahren der Prokaryonten
Introns existiert haben, diese allerdings durch den hohen
Selektionsdruck, sich schnell zu replizieren eine ,,Ballast``
darstellten, die mit der Zeit verloren ging.
Man nimmt an, das der Verlust von Introns auf die Aktivität einer
Reversen Transkriptase (z.B, aus einem Retrovirus) zurückzuführen
ist. Diese würde eine RNA in
eine DNA umschreiben und bei einer Rekombination dieser DNA mit der
genomischen DNA käme es dann zu einer Elimination des Introns.
Wenn man die gesamte DNA des Menschend denaturiert, in ca. 1000 bp
lange Stücke schneidet und dann unter
stringenten Bedingungen wieder renaturiert, finden sich einige
Sequenzen, die auf Grund von ihrem häufigen Vorkommen schneller
hybridisieren als andere.
Bei einigen dieser Sequenzen handelt es sich um Transposons, die sich
mehrfach in das Genom kopiert haben, bei anderem um sogenannte
Satelliten-DNA.
Die letzteren bestehen aus einer sehr langen Tandem-Wiederholung einer sehr
kurzen Sequenz. Diesen Sequenzen konnte man noch keine Funktion
zuordnen. Sie verändern ihre Postition sehr schnell und man vermutet,
dass ihr einziger Zweck darin liegt, das sie ihre eigene Präsenz
erhalten. Man nennt sie deshalb auch ,,selbstische DNA``.
Die transponierbaren Elemente können sich entweder direkt als
DNA-Fragmente im Genom bewegen oder sie tun dies über die
Zwischenstufe einer RNA-Form. Sie machen bei höheren Eukaryonten
ca. 10% des Genoms aus.
Die meisten aller Transponierbaren Elemente lassen sich entweder den
L1-transponierbaren Elementen, die sich mit einer Reversen
Transcriptase bewegen und die sehr kurze Alu-Sequenz, die sich wie ein
transponierbares Element bewegt unterteilen.
Sie sind für viele Spontanmutationen
verantwortlich, da sie bei ihrer Insertion und Deletion eine
Zielstellenduplikation durchführen, bei der die Base an der die
Stelle, an der das Element eingefüg wurde dupliziert oder deletiert
wird.
Häufig entstehen aus den Transposons sogenannte Enhancer, die die
Expression eines Genes verändern. Die Wanderung dieser Verstärker
könnte eine Langzeitoptimierung der Genexpression zur Folge haben.
Das Vorhandensein der Nukleosomen behindert die Regulationsproteine,
wie auch die Polymerase selbst.
Einige DNA-Stücke sind zu steif, um sich zu Nukleosomen zu formen. An
diesen freien Regionen können die Proteine ungehindert binden.
Wenn die Proteine im Bereich des Nukleosoms binden, dann
destabilisieren sie es und führen dazu, dass es sich auflöst. Die
allgemeinen Transkriptionsfaktoren können hingegen ohne dass ein
Regulatorprotein gebunden ist, keine Transkriütion initiieren. Dies
verhindert einen Transkriptionsstart ohne gebunden Aktivatorproteine.
An einigen Stellen ist das Chromatin zu einem besonders dichten
Heterochromatin verpackt und lässt keine Transkription der Gene
zu. Dies wird Silencing genannt.
Beim Säuger gibt es ein Cluster von Globin-Genen, das durch eine
dichte Chromatinpackung deaktiviert werden kann. Dieser Vorgang wird
von einer Locus-Kontrolltregion (LCR) kontrolliert. Der molekulare
Mechanismus dieser Kontrolle ist jedoch noch nicht bekannt.
Hypothetische Mechanismen sind ein wanderndes Protein, das am
Chromatin entlangwandert und die Packung
aktiv auflockert, ein Enyzm, das das unter Spannung stehende Chromatin
,,entspiralisiert`` oder Proteine, die von allen Seiten her
die dichte Packungsstruktur auflösen.
Bei dem Bacterium Salmonelle findet man einen
Differenzierungsmechanismus, der als Phasen-Variation bezeichnet wird
und auf der Inversion eines DNA-Stücks von 1000 bp Länge beruht.
Mit der unterschiedlichen Orientierung dieses Promotors wird ein
unterschiedliches Oberflächenprotein synthetisiert. Dies dient sehr
wahrscheinlich als Abwehrmechanismus gegen das Immunsystem des
Wirtes.
Bei der Bäckerhefe können sich zwei haploide Zellen, wenn sie einen
unterschiedlichen Paarungstyp (a und 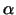 ) aufweisen, miteinander
zu einer diploiden Zelle vereinigen. Diese kann sich dann nicht mehr
paaren, ist dafür aber in der Lage, in schlechten Zeiten Sporen
auszubilden. ) aufweisen, miteinander
zu einer diploiden Zelle vereinigen. Diese kann sich dann nicht mehr
paaren, ist dafür aber in der Lage, in schlechten Zeiten Sporen
auszubilden.
Der Zelltyp a synthetisiert nur ein Regulatorprotein a1, das in der
haploiden Zelle ohne Wirkung ist. In der 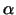 -Zelle werden zwei
Regulationsproteine -Zelle werden zwei
Regulationsproteine 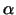 1 und 1 und 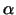 2 synthetisiert. Das 2 synthetisiert. Das
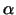 2-Protein dient als Repressor, der die a-Gene ausschaltet. 2-Protein dient als Repressor, der die a-Gene ausschaltet.
Wenn nun die beiden zellen fusionieren, führt eine kombinatorische
Kontrolle der beiden Faktoren a1 und 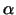 2 dazu, dass ein neues,
für die diploiden Zellen typisches Gen-Set eingeschaltet wird. 2 dazu, dass ein neues,
für die diploiden Zellen typisches Gen-Set eingeschaltet wird.
Es ist bei einigen Stämmen möglich, dass sich der a- in den
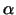 -Typ umwandelt, indem eine unterschiedliche Genkassette an
einen anderen Locus transferiert wird. -Typ umwandelt, indem eine unterschiedliche Genkassette an
einen anderen Locus transferiert wird.
Bei dem Bakteriophagen lambda findet man ein anderes wichtiges Prinzip
der Genregulation, bei dem zwei Proteine ihre Synthese gegenseitig
unterdrücken. Dies führt zu einem binären Schalter, der bestimmt, ob
sich der Phage zusammen mit dem Bakteriengenom vermehrt oder ob er in
den lytischen Zustand übergeht.
|





