Unterabschnitte
Proteine
Proteinstruktur
Die meisten Makromoleküle bilden eine Struktur, die neben der
kovalenten Bindung der Monomere auf folgende Bindungstypen stützt:
- Wasserstoffbrückenbindungen
- van der Waals-Kräfte
- Ionenbindungen
Die Proteine bilen ihre Struktur vor allem durch Wechselwirkungen im
polaren Milieu, ausserdem findet man - vor allem bei sezernierten und
Oberflächenproteinen - auch Disulfidbrücken.
Die zwei strukturgebenden Motive der Proteine sind die 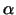 -Helix
und das -Helix
und das 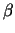 -Faltblatt. Die -Faltblatt. Die 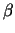 -Faltblattstrukturen findet man
vor allem im Kern der meisten Proteine. -Faltblattstrukturen findet man
vor allem im Kern der meisten Proteine.
Die Helices bilden sich durch Wasserstoffbrücken zwischen der C=O und
der NH-Gruppe mit jeweils vier Aminosäuren Abstand.
Die Faltblätter bilden sich in Abwesenheit von Alanin und Glycin durch
das Aneinanderlagern von mehreren Ketten.
Die 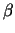 -Faltblätter bilden
dort eine Stützkonstruktion für die übrige Struktur. -Faltblätter bilden
dort eine Stützkonstruktion für die übrige Struktur.
Mehrere 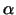 -Helices können sich zu coiled coil -
Doppelwechselstrukturen zusammenlagern und sind so auch in wässrigen
Milieu aktiv. -Helices können sich zu coiled coil -
Doppelwechselstrukturen zusammenlagern und sind so auch in wässrigen
Milieu aktiv.
Die Struktur wird in folgende Ebenen gegliedert:
- Die Primärstruktur eines Proteins ist die Sequenz der
Aminosäuren.
- Die Sekundärstruktur von
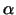 -Helices und -Helices und
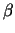 -Faltblättern bildet sich automatisch. Kombinationenen aus
Faltblättern und Helices bilden sogenannte Domänen aus. -Faltblättern bildet sich automatisch. Kombinationenen aus
Faltblättern und Helices bilden sogenannte Domänen aus.
- Die Tertiärstruktur ist die dreidimensionale Anordnung
des Proteins.
- Die Quatermärstrukturen bilden sich durch die Bildung
von Dimeren oder Oligomeren.
Nur aus relativ wenigen Kombinationen von Aminosäuren ergeben sich
sinnvolle Proteine, weshalb neue Proteine häufig aus bestehenden
Stukturen durch Modifikation hervorgehen.
Häufig bilden mehrere globuläre Proteine einen Proteinkomplex. So
können mehrere Polypeptide zu neuen Strukturen zusammengefügt
werden. Im Laufe der Evolution wurden häufig die DNA-Domänen solcher
Komplexe zusammengefasst und zu einem Gen, das eine grosse
Polypeptidkette kodiert verknüpft. Heute sind diese Vorgänge an den
Multidomänstruktuden der Proteine zu erkennen.
Das Zusammensetzen mehrere Einzelstrukturen findet man auch bei
grossen Strukturen wie den Ribosomen.
Wenn eine Untereinheit eine Bindungsstelle für das eigene Protein
besitzt, so kann es im einfachsten Fall Dimere oder wenn die
Bindungsstelle ausserhalb der Erkennungsregion liegt auch eine Kette
bilden (Aktin).
Durch die Verbindung von mehreren Strukturen zu Doppelwendeln oder wie
im Fall vieler einfacher Virushüllen zu einer Hohlkugel verleihen den
Proteinen eine zusätzliche Stabilität. Viele dieser Aggregate können
sich auch in vitro bilden (Tabakmosaikvirus oder Bakterienribosom),
während andere Aggregate sich nur dann bilden, wenn andere Enzyme
vorhanden sind (proteolytische Synthes von Insulin aus Proinsulin).
Der Aufbau komplexer Strukturen aus vorgefertigten Untereinheiten hat
mehrere Vorteile: Die Synthese und das Auseinandernehmen benötigt, auf
Grund der nichtkovalenten Bindungen wenig Energie, Fehler werden
minimiert, das mutierte Untereinheiten aussortiert werden können und
die Verwendung einer Untereinheit in mehreren Komplexen verringert die
Menge an benötigter genetischer Information.
Für die Zelle ist die Mutation bekannter Strukturen immer der
ökonomischere Weg als komplette ,,Neuentwicklung`` von
Strukturen durch Zufallsmutationen. In der Praxis kann man dies
ausnutzen, indem man die Aminosäuresequenzen neuer mit denen bekannter
Proteine vergleicht und so durch Sequenzhomologien die Struktur und
Funktion des unbekannten Proteins vermuten kann.
Proteinfaltung
Die Primärstruktur enthält alle Informationen zur Bildung der
Sekundärstruktur. Ein weiteres Einwirken anderer Proteine ist nicht
notwendig. Man kann dies durch Denaturieren von Proteinen in vitro
zeigen: Diese renaturieren wieder.
Damit ein Protein die richtige Konformation (Tertiärstruktur)
einnimmt, sind teilweise Cofaktoren oder Untereinheiten erforderlich.
Dies können entweder eine stöchiometrische Wirkung haben (Chaperone
verhindern durch ihre Anlagerung eine falsche Faltung) oder sie haben
eine katalytische Wirkung, indem sie Disulfidbrücken bilden
(z.B. Proteindisulfidisomerase) oder eine Bindungsänderung von cis
nach trans durchführen (Peptidylpropylisomerase).
Eine Modifikation der Aminosäuren kann ebenfalls die Konformation
ändern. Diese Modifikationen können Phosphorylierungen,
Methylierungen, Acetylierungen oder Glycosylierungen sein.
Protein finden sich - auch nach Denaturierung - relativ schnell in
einer sehr offenen und flexiblen Form, dem Molten Globule wieder. Die
eigentliche Faltung wird jedoch unter der Kontrolle molekularer
Chaperone durchgeführt. Die Chaperone sind mit den
Hitzeschockproteinen verwandt und helfen den Proteinen aktiv bei der
Proteinfalung oder verändern deren Struktur, wenn sie sich zunächst
falsch gefaltet haben.
Einzelne Domänen eines Proteins falten sich gleich zu Anfang in einer
charakteristischen Form. Sie werden Moduln genannt. Bei einer
Tandemduplikation können diese Moduln leicht mehrfach in ein Protein
eingebaut werden.
Durch unterschiedliche Mechanismen können unterschiedliche Proteine
miteinander in Wechselwirkung treten:
Eine Möglichkeit besteht darin, dass das eine Protein eine SH2-Domäne
aufweist und darin das phosphorylierte Tyrosin eines anderen Proteins
bindet. Hierbei lagert sich eine ,,Strippe`` mit einer
Oberfläche zusammen.
Eine andere Möglichkeit besteht in der Zusammenlagerung zweier
helicaler Strukturen.
Die dritte Möglichkeit iest das Zusammengehen zweier Oberflächen durch
schwache Bindungen.
Der Zusammenbau grosser Proteinkomplexe wird druch zwei Mechanismen
kontrolliert: Zum einen wird nach dem in
2.2.5 vorgestellten Mechanismus der
Kopplung das Binden den zweiten Partners durch den ersten gefördert,
aussdem werden unvollständige Proteinkomlexe durch Proteasen
abgebaut.
Proteinabbau
Wenn ein Protein abgebaut werden soll, wird ein kleines Protein - das
Ubiquitin - an das Protein geknüpft. Dies geschieht durch spezielle
Enzyme, die eine im Normalzustand im Inneren liegende Aminosäurekette
erkennen und markieren.
So wird dieses von den
Proteasomen erkannt und in deren zylinderartigen Inneren abgebaut.
Nach der N-Ende-Regel besteht ein enger Zusammenhang zwischen der
Halbwertszeit eines Proteins in vivo und dessen N-terminaler
Aminosäure.
Spezielle Aminosäuren bilden ein Abbausignal, das unter anderem
eventuell verhindern soll, dass Proteine, die eigentlich in der
Membran lokalisiert sein sollten, ins Cytosol wandern.
Durch eine Acetylierung kann der Abbau auch bei einer
destabilisierenden Aminosäure verhindert werden.
Proteinfunktion
Damit ein Protein mit seinem Liganden interagieren kann, müssen sich
zwischen den beiden Partnern schwache nichtkovaltene Bindungen
ausbilden. Die Inteaktion wird durch die Reaktion bestimmter
Aminosäuren mit dem Substrat erreicht. Wenn die Reaktivität einer
solchen Seitenkette nicht ausreicht, dann werden nicht-polypeptidische
Coenzyme (teilweise sehr fest) an das Protein gebunden (z.B. Häm) auch
nach der Bindung an das Protein noch eine Reaktivität bewahren und
mittels dieser zu einer Substratspezifität (Sauerstoff) führen.
Normalerweise bindet das Enzym zunächst das Substrat, wandelt es in
ein Produkt um und entlässt dieses aus der Bindung. Da alle drei
Reaktionen Gleichgewichtsreaktionen sind, wird die Reaktion durch
eine Erhöhung der Substratrate bis zu einer Sättigung Vmax
gesteigert. Der Substratwert, der der Reaktionsgeschwindigkeit bei der
Hälfte von Vmax entspricht wird Michaelis-Konstante KM
genannt.
Enzyme katalysieren eine Reaktion, indem bestimmte Übergangszustände
stabilitiert und die Elektronenkonfiguration des so gebundenen
Moleküls verändert werden. Eine andere Wirkung besteht darin, ein
Substrat kurzzeitig zu binden, es zur Reaktion zu bringen, um dann die
kovalenten Bindung wieder zu lösen.
Allerdings beschleunigt ein Enyzm immer die Hin- und die Rückreaktion
in gleichem Masse. Das Verhältnis von Hin- und Rückreaktion hängt nur
von deren Konzentrationen ab. Energetisch ungünstige Reaktinen laufen
durch eine Koppelung mit ATP-Hydrolyse ab. Durch dieses Prozesse wird
der gesamte ATP-Vorrat einer Zelle innerhalb von 1-2 Minuten komplett
umgesetzt.
Normalerweise sind alle Reaktionen diffusionsbegrenzt, d.h. dass sie
immer nur dann ablaufen, wenn die beiden Substrate
zusammentreffen. Dieses Manko kann durch die Bildung von
Multienzymkomplexen, bei denen das Produkt des einen Moleküls an das
nächste als Substrat weiter gereicht wird, verhindert werden.
Regulation der Proteinfunktion
Durch die Bindung eines Liganden verändert sich die Struktur des
Proteins. Häufig wechselt das Protein von einer offenen in eine
geschlossene Konformation, indem sich zwei Proteindomänen aufeinander
zu bewegen. In der veränderte Konformation kann dann das Protein ein
anderes Substrat (z.B. ATP) besser binden und so kommt es dann zur
Reaktion.
Wenn ein weiteres Substrat an einer anderen Bindungsstelle des Proteins binden
kann und dadurch die Konformation verändert, kann es zu einer
kooperativen oder einer kompetetiven Bindung kommen. Eine solche
Kopplung wird Allosterie genannant. So ist es möglich, dass eine
Substanz die Bindung zwischen einem Enyzm und seinem Substrat entweder
fördern oder Hemmen.
Wichtig ist dabei dass die Hemmunf oder Förderung an einer Stelle
erfolgt, die sich vom katalytischen Zentrum des Enzyms unterscheidet.
Bei einer längerern Reaktionkette findet man häufig eine negative
Rückkopplung, bei der das Endprodukt der Reaktionskette das ersten
Enyzm der Kette hemmt. Das Endprodukt bindet an das Regulationszentrum
des Enzyms, während das Startprodukt am Aktivitätzentrum bindet.
Bei einer positiven Rückkopplung hingegen aktiviert der Ligand das
Enzym durch Binden an die inaktivierte Form. Solche
Kopplungsmechanismen dienen der Kontrolle kataboler Prozesse, bei
denen beispielsweise das ADP in genügender Menge die Reaktion
aktiviert, was zur Bildung von ATP führt.
Die meisten allosterischen Mechanismen wirken an Enzymen mit mehreren
Untereinheiten. Der Ligand bindet an eine der Untereinheiten, wodurch
diese die anderen Untereinheiten so verändert, dass es zu einem
gemeinsamen kooperativen Übergang kommt, der eine sigmoide Kurve nach
dem Alles-oder-Nichts-Prinzip zur Folge hat.
Eine andere Möglichkeit der Beeinflussung einer Enzyms besteht in der
Phosphorylierung einer Seitengruppe durch eine Proteinkinase, was
durch die zweifach negative
Ladung der Phosphatgruppe eine Konformationsänderung zur Folge haben
kann. Auch hier liegt die Phosphorylierungsstelle meist ausserhalb des
aktiven Zentrums und beeinflusst das Enzym allosterisch.
Während die Proteinkinasen jeweils eine Phosphatgruppe vom ATP auf das
Protein übertragen, entfernen die Protein-Phosphatasen die
Phosphatgruppe. In beiden Enzymgruppen gibt es Enyzme mit
unerschiedlichem Spezigitätsgrad.
Häufig findet man Enzyme, die nur dann aktiv werden, wenn mehrere
,,Bedingungen`` erfüllt sind. So wird die Cyklin-abhängige
Proteinkinase nur dann aktiv, wenn ein bestimmtes Phosphat entfernt,
ein anderes hinzugefügt und Cyclin gebunden ist. So können sehr
komplexe Kontrollmechanismen realisiert werden.
Bei den Eukaryotenzellen findet man häufig GTP-bindende Proteine, die
nur in der Anwesenheit von GTP aktiv sind. Das Protein bindet das GTP,
wird dadurch aktiviert und inaktiviert sich selbst anschliessend durch
Hydrolyse des GTP zu GDP.
Die Regulation der GTP-bindenden Proteine erfolgt durch
GTPase-aktivierende Proteine kontrolliert. Diese binden an das
GTP-bindende Protein und veranlassen es zur GTP-Hydrolyse. Trifft das
inaktivierte Protein dann auf ein Guanin-Nucleotid-freisetzendes
Protein (GNRP), so setzt es das GDP frei, bindet ein GTP und wird
wieder aktiv.
Bei einem Motorprotein, das sich an einer Struktur entlangbewegt, ist
es wichtig, dass die Bewegung gerichtet geschieht. Dies wird durch die
Verwendung der ATP-Hydrolyse erreicht. Die Hydrolyse ist energetisch
ein praktisch irreversibler Prozess, der dazu führt, dass
Konformationsänderungen gerichtet ablaufen.
In diesem Fall kann man sich dies so vorstellen, dass ein Enzym zwei
,,Füsse`` besitzt, von denen einer Kontakt mit der Stuktur
hat. Durch die ATP-Bindung nimmt der zweite Fuss Kontakt mit der
Struktur auf. Durch die Hydrolyse wird der zweite Fuss nachgezogen und
nachdem sich das ADP gelöst hat ist die Ausgangskonfiguration wieder
erreicht. Durch den Hydrolyseschritt findet die Reaktion in
umgekehrter Reihenfolge nicht statt.
Ein solcher Ablauf führt z.B. beim Myosin dazu, dass der Kopf seine
Bewegung macht.
Ein allosterisches Protein in der Membran kann entweder und
ATP-Hydrolyse einen Ionengradienten aufbauen oder umgekehrt durch
einen Ionengradienten ATP synthetisieren.
Viele andere Prozesse in der Zelle laufen nach ähnlichen Mechanismen
ab: Motoren, Zeitgeber, Montagefaktoren und Informationsüberträger.
|





